Die vorliegende Übersetzung wurde maschinell erstellt. Im Falle eines Konflikts oder eines Widerspruchs zwischen dieser übersetzten Fassung und der englischen Fassung (einschließlich infolge von Verzögerungen bei der Übersetzung) ist die englische Fassung maßgeblich.
Anwendungsfall: Entwicklung einer Anwendung für medizinische Intelligenz mit erweiterten Patientendaten
Generative KI kann dazu beitragen, die Patientenversorgung und die Produktivität des Personals zu verbessern, indem sowohl die klinischen als auch die administrativen Funktionen verbessert werden. KI-gestützte Bildanalysen, wie z. B. die Interpretation von Sonogrammen, beschleunigen die Diagnoseprozesse und verbessern die Genauigkeit. Sie kann wichtige Erkenntnisse liefern, die rechtzeitige medizinische Interventionen unterstützen.
Wenn Sie generative KI-Modelle mit Wissensgraphen kombinieren, können Sie die chronologische Organisation elektronischer Patientenakten automatisieren. Auf diese Weise können Sie Echtzeitdaten aus Interaktionen, Symptomen, Diagnosen, Laborergebnissen und Bildanalysen zwischen Arzt und Patient integrieren. Dadurch erhält der Arzt umfassende Patientendaten. Diese Daten helfen dem Arzt, genauere und zeitnahere medizinische Entscheidungen zu treffen, was sowohl die Behandlungsergebnisse als auch die Produktivität der Gesundheitsdienstleister verbessert.
Übersicht über die Lösung
KI kann Ärzte und Kliniker unterstützen, indem sie Patientendaten und medizinisches Wissen zusammenführt, um wertvolle Erkenntnisse zu gewinnen. Bei dieser Retrieval Augmented Generation (RAG) -Lösung handelt es sich um eine Engine für medizinische Intelligenz, die umfassende Patientendaten und Erkenntnisse aus Millionen von klinischen Interaktionen nutzt. Sie nutzt die Leistungsfähigkeit der generativen KI, um evidenzbasierte Erkenntnisse für eine verbesserte Patientenversorgung zu gewinnen. Es wurde entwickelt, um die klinischen Arbeitsabläufe zu verbessern, Fehler zu reduzieren und die Behandlungsergebnisse zu verbessern.
Die Lösung umfasst eine automatisierte Bildverarbeitungsfunktion, die von unterstützt wird. LLMs Diese Funktion reduziert den Zeitaufwand, den das medizinische Personal für die manuelle Suche nach ähnlichen Diagnosebildern und die Analyse der Diagnoseergebnisse aufwenden muss.
Die folgende Abbildung zeigt die Lösung end-to-end-workflow für diese Lösung. Es verwendet Amazon Neptune, Amazon SageMaker AI, Amazon OpenSearch Service und ein Basismodell in Amazon Bedrock. Für den Context Retrieval Agent, der mit dem Medical Knowledge Graph in Neptune interagiert, können Sie zwischen einem Amazon Bedrock-Agenten und einem LangChain Agent.
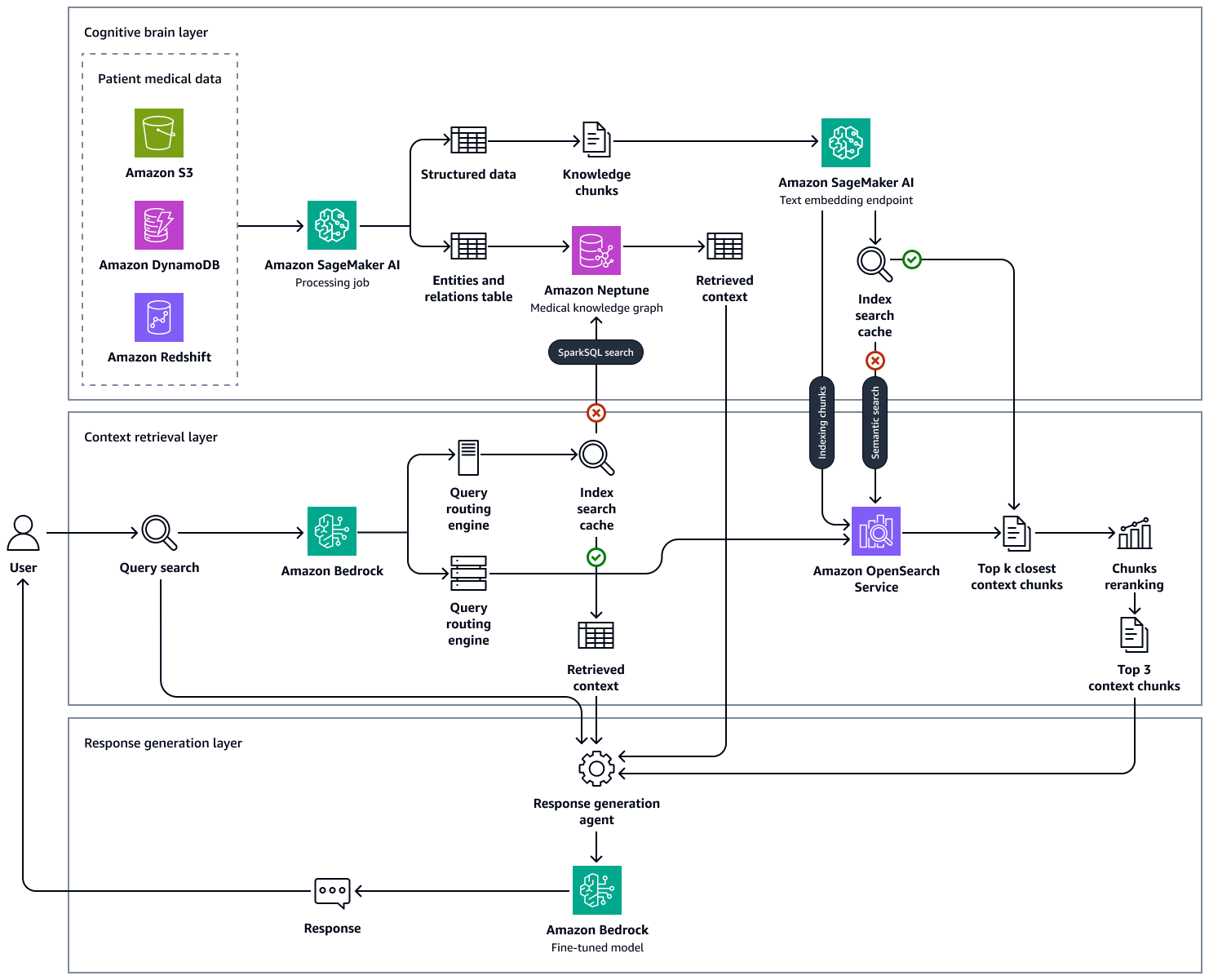
In unseren Experimenten mit medizinischen Musterfragen stellten wir fest, dass die endgültigen Antworten, die durch unseren Ansatz mithilfe eines in Neptune verwalteten Wissensgraphen, einer OpenSearch Vektordatenbank mit klinischer Wissensdatenbank und Amazon Bedrock generiert LLMs wurden, auf Fakten beruhten und weitaus genauer sind, da die falsch positiven Ergebnisse reduziert und die wahren positiven Ergebnisse verstärkt werden. Diese Lösung kann evidenzbasierte Erkenntnisse über den Gesundheitszustand der Patienten liefern und zielt darauf ab, die klinischen Arbeitsabläufe zu verbessern, Fehler zu reduzieren und die Behandlungsergebnisse zu verbessern.
Der Aufbau dieser Lösung besteht aus den folgenden Schritten:
Schritt 1: Daten ermitteln
Es gibt viele medizinische Open-Source-Datensätze, die Sie verwenden können, um die Entwicklung einer KI-gestützten Lösung für das Gesundheitswesen zu unterstützen. Ein solcher Datensatz ist der MIMIC-IV-Datensatz
Sie können auch einen Datensatz verwenden, der kommentierte, anonymisierte Zusammenfassungen von Patientenentlassungen enthält, die speziell für Forschungszwecke zusammengestellt wurden. Ein Datensatz mit einer Zusammenfassung der Entlassung kann Ihnen beim Experimentieren mit der Extraktion von Entitäten helfen, sodass Sie wichtige medizinische Entitäten (wie Erkrankungen, Verfahren und Medikamente) anhand des Textes identifizieren können. Schritt 2: Erstellung eines medizinischen WissensgraphenIn diesem Leitfaden wird beschrieben, wie Sie die strukturierten Daten aus den Datensätzen MIMIC-IV und Zusammenfassung der Entlassungsdaten verwenden können, um ein medizinisches Wissensdiagramm zu erstellen. Dieser Graph zum medizinischen Wissen dient als Grundlage für fortschrittliche Abfrage- und Entscheidungsunterstützungssysteme für medizinisches Fachpersonal.
Zusätzlich zu textbasierten Datensätzen können Sie Bilddatensätze verwenden. Zum Beispiel der Datensatz Musculoskeletal Radiographs (MURA), bei dem es sich um eine
Schritt 2: Erstellung eines medizinischen Wissensgraphen
Für jede Gesundheitsorganisation, die ein System zur Entscheidungsunterstützung aufbauen möchte, das auf einer riesigen Wissensbasis basiert, besteht eine zentrale Herausforderung darin, die medizinischen Entitäten zu finden und zu extrahieren, die in den klinischen Aufzeichnungen, medizinischen Fachzeitschriften, Entlassungszusammenfassungen und anderen Datenquellen enthalten sind. Sie müssen auch die zeitlichen Zusammenhänge, Themen und Sicherheitseinschätzungen aus diesen Krankenakten erfassen, um die extrahierten Entitäten, Attribute und Beziehungen effektiv nutzen zu können.
Der erste Schritt besteht darin, medizinische Konzepte aus dem unstrukturierten medizinischen Text zu extrahieren, indem ein paar Eingabeaufforderungen für ein Basismodell verwendet werden, wie Llama 3 in Amazon Bedrock. Beim Few-Shot-Prompting stellen Sie einem LLM eine kleine Anzahl von Beispielen zur Verfügung, die die Aufgabe und das gewünschte Ergebnis demonstrieren, bevor Sie es bitten, eine ähnliche Aufgabe auszuführen. Mithilfe eines LLM-basierten Extraktors für medizinische Entitäten können Sie den unstrukturierten medizinischen Text analysieren und anschließend eine strukturierte Datendarstellung der medizinischen Wissenseinheiten generieren. Sie können die Patientenattribute auch für nachgelagerte Analysen und Automatisierung speichern. Der Vorgang zur Extraktion von Entitäten umfasst die folgenden Aktionen:
-
Extrahieren Sie Informationen über medizinische Konzepte wie Krankheiten, Medikamente, Medizinprodukte, Dosierung, Häufigkeit und Dauer der Behandlung, Symptome, medizinische Eingriffe und deren klinisch relevante Eigenschaften.
-
Erfassen Sie funktionale Merkmale, wie z. B. zeitliche Beziehungen zwischen extrahierten Entitäten, Subjekten und Sicherheitsbewertungen.
-
Erweitern Sie medizinische Standardvokabeln, wie z. B. die folgenden:
-
Codes aus der Internationalen Klassifikation der Krankheiten, 10. Revision, klinische Modifikation
(ICD-10-CM) -
Begriffe aus medizinischen Fachüberschriften
(MeSH) -
Konzepte aus der Systematisierten Nomenklatur der Medizin,
klinische Begriffe (SNOMED CT) -
Codes aus dem Unified
Medical Language System (UMLS)
-
Fassen Sie Entlassungsbescheinigungen zusammen und leiten Sie medizinische Erkenntnisse aus Zeugnissen ab.
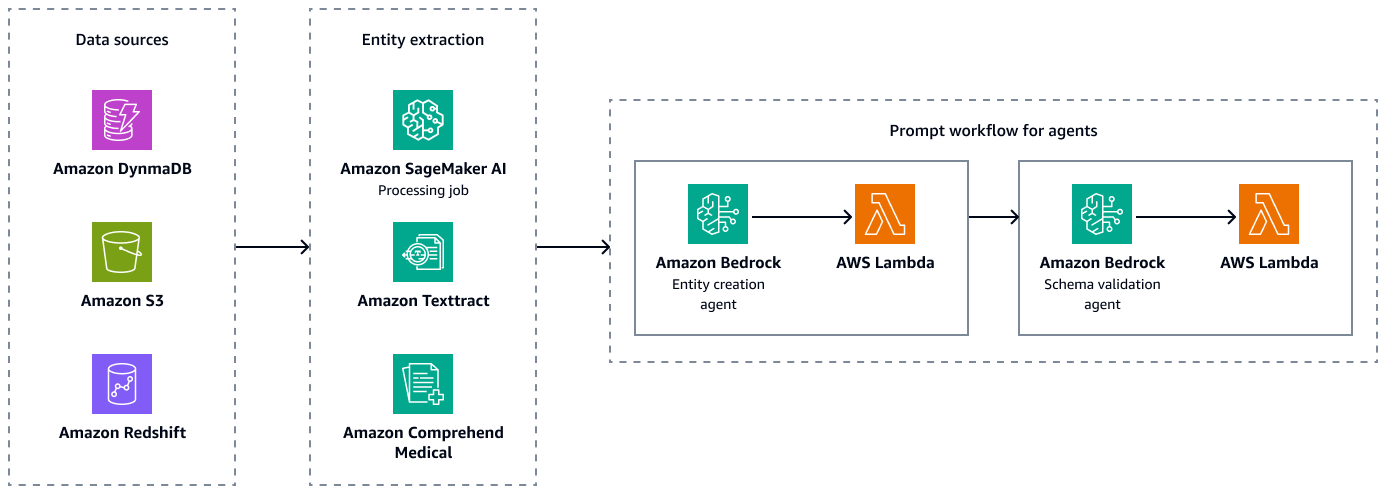
Die folgende Abbildung zeigt die Schritte zur Entitätsextraktion und Schemavalidierung, um gültige Kombinationen von Entitäten, Attributen und Beziehungen zu erstellen. Sie können unstrukturierte Daten wie Entlassungszusammenfassungen oder Patientennotizen in Amazon Simple Storage Service (Amazon S3) speichern. Sie können strukturierte Daten wie ERP-Daten (Enterprise Resource Planning), elektronische Patientenakten und Laborinformationssysteme in Amazon Redshift und Amazon DynamoDB speichern. Sie können einen Amazon Bedrock Entity Creation Agent erstellen. Dieser Agent kann Dienste wie Amazon SageMaker AI-Datenextraktionspipelines, Amazon Textract und Amazon Comprehend Medical integrieren, um Entitäten, Beziehungen und Attribute aus den strukturierten und unstrukturierten Datenquellen zu extrahieren. Schließlich verwenden Sie einen Amazon Bedrock-Schemavalidierungsagenten, um sicherzustellen, dass die extrahierten Entitäten und Beziehungen dem vordefinierten Diagrammschema entsprechen, und um die Integrität der Knoten-Edge-Verbindungen und der zugehörigen Eigenschaften aufrechtzuerhalten.
Nach der Extraktion und Validierung der Entitäten, Beziehungen und Attribute können Sie sie verknüpfen, um ein subject-object-predicate Triplett zu erstellen. Sie nehmen diese Daten in eine Amazon Neptune Neptune-Graphdatenbank auf, wie in der folgenden Abbildung dargestellt. Graphdatenbanken sind für das Speichern und Abfragen der Beziehungen zwischen Datenelementen optimiert.
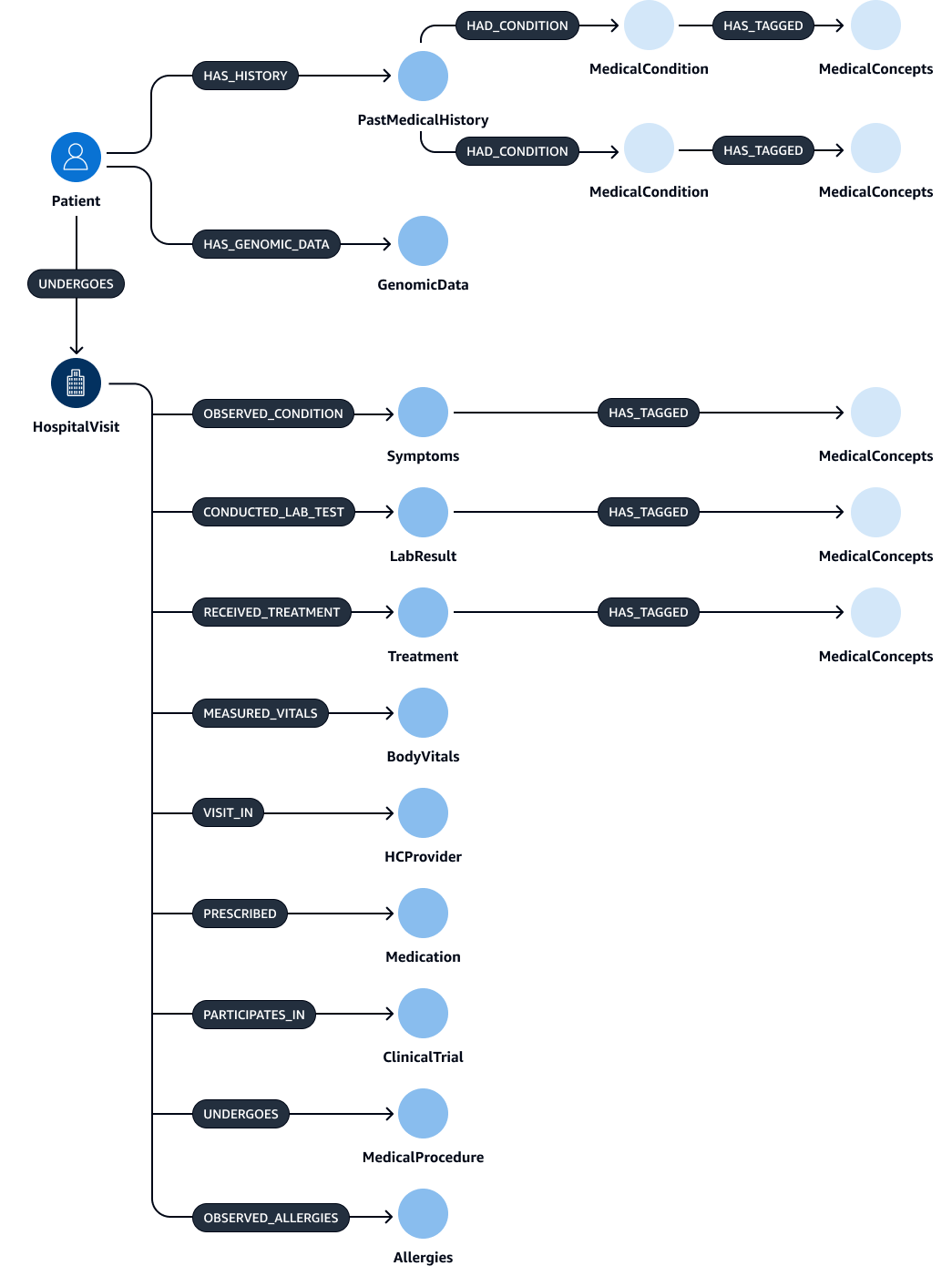
Sie könnten mit diesen Daten einen umfassenden Wissensgraphen erstellen. Ein WissensgraphHospitalVisit,PastMedicalHistory, SymptomsMedication,MedicalProcedures, undTreatment.
In den folgenden Tabellen sind die Entitäten und ihre Attribute aufgeführt, die Sie aus Entlassungsnotizen extrahieren können.
| Entität | Attribute |
|---|---|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
In der folgenden Tabelle sind die Beziehungen aufgeführt, die möglicherweise zwischen Entitäten bestehen, und die entsprechenden Attribute. Beispielsweise könnte die Patient Entität eine Verbindung zu der HospitalVisit Entität mit der [UNDERGOES] Beziehung herstellen. Das Attribut für diese Beziehung istVisitDate.
| Betreff-Entität | Beziehung | Objekt-Entität | Attribute |
|---|---|---|---|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
Keine |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
Keine |
|
|
|
Keine |
|
|
|
Keine |
|
|
|
Keine |
Schritt 3: Erstellung von Context-Retrieval-Agenten zur Abfrage des medizinischen Wissensgraphen
Nachdem Sie die medizinische Graphdatenbank erstellt haben, besteht der nächste Schritt darin, Agenten für die Graphinteraktion zu erstellen. Die Agenten rufen den richtigen und erforderlichen Kontext für die Abfrage ab, die ein Arzt oder Kliniker eingibt. Es gibt mehrere Optionen für die Konfiguration dieser Agenten, die den Kontext aus dem Knowledge Graph abrufen:
Amazon Bedrock-Agenten für die Interaktion mit Diagrammen
Amazon Bedrock-Agenten arbeiten nahtlos mit Amazon Neptune Neptune-Graphdatenbanken zusammen. Sie können erweiterte Interaktionen über Amazon Bedrock-Aktionsgruppen durchführen. Die Aktionsgruppe initiiert den Prozess, indem sie eine AWS Lambda Funktion aufruft, die Neptune OpenCypher-Abfragen ausführt.
Für die Abfrage eines Wissensgraphen können Sie zwei unterschiedliche Ansätze verwenden: direkte Abfrageausführung oder Abfragen mit Kontexteinbettung. Diese Ansätze können unabhängig voneinander oder kombiniert angewendet werden, abhängig von Ihrem spezifischen Anwendungsfall und Ihren Rangkriterien. Durch die Kombination beider Ansätze können Sie dem LLM einen umfassenderen Kontext bieten, was die Ergebnisse verbessern kann. Im Folgenden sind die beiden Ansätze zur Abfrageausführung aufgeführt:
-
Direkte Ausführung von Cypher-Abfragen ohne Einbettungen — Die Lambda-Funktion führt Abfragen direkt gegen Neptune aus, ohne dass eine auf Einbettungen basierende Suche erforderlich ist. Im Folgenden finden Sie ein Beispiel für diesen Ansatz:
MATCH (p:Patient)-[u:UNDERGOES]->(h:HospitalVisit) WHERE h.Reason = 'Acute Diabetes' AND date(u.VisitDate) > date('2024-01-01') RETURN p.PatientID, p.Name, p.Age, p.Gender, p.Address, p.ContactInformation -
Direkte Ausführung von Cypher-Abfragen mithilfe der eingebetteten Suche — Die Lambda-Funktion verwendet die eingebettete Suche, um die Abfrageergebnisse zu verbessern. Dieser Ansatz verbessert die Abfrageausführung durch die Integration von Einbettungen, bei denen es sich um dichte Vektordarstellungen von Daten handelt. Einbettungen sind besonders nützlich, wenn für die Abfrage semantische Ähnlichkeit oder ein umfassenderes Verständnis erforderlich ist, das über exakte Übereinstimmungen hinausgeht. Sie können vortrainierte oder individuell trainierte Modelle verwenden, um Einbettungen für jede Erkrankung zu generieren. Im Folgenden finden Sie ein Beispiel für diesen Ansatz:
CALL { WITH "Acute Diabetes" AS query_term RETURN search_embedding(query_term) AS similar_reasons } MATCH (p:Patient)-[u:UNDERGOES]->(h:HospitalVisit) WHERE h.Reason IN similar reasons AND date(u.VisitDate) > date('2024-01-01') RETURN p.PatientID, p.Name, p.Age, p.Gender, p.Address, p.ContactInformationIn diesem Beispiel ruft die
search_embedding("Acute Diabetes")Funktion Erkrankungen ab, die dem Begriff „Akuter Diabetes“ semantisch nahe kommen. Auf diese Weise kann die Abfrage auch nach Patienten mit Erkrankungen wie Prädiabetes oder metabolischem Syndrom suchen.
Die folgende Abbildung zeigt, wie Amazon Bedrock-Agenten mit Amazon Neptune interagieren, um eine Cypher-Abfrage eines medizinischen Wissensgraphen durchzuführen.
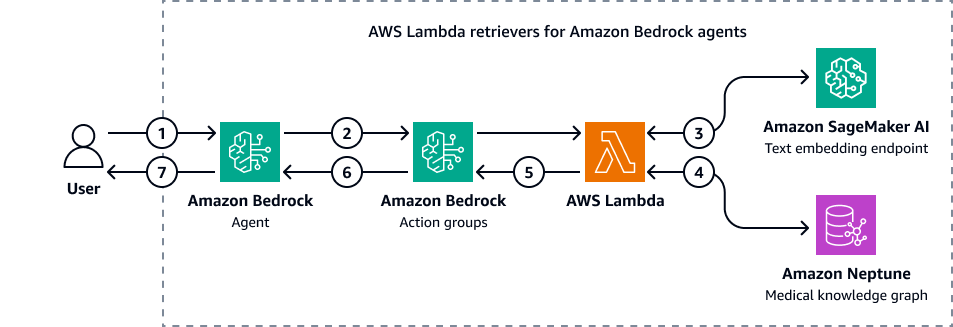
Das Diagramm zeigt den folgenden Workflow:
-
Der Benutzer sendet eine Frage an den Amazon Bedrock-Agenten.
-
Der Amazon Bedrock-Agent leitet die Frage und die Eingabefiltervariablen an die Amazon Bedrock-Aktionsgruppen weiter. Diese Aktionsgruppen enthalten eine AWS Lambda Funktion, die mit dem Amazon SageMaker AI-Endpunkt zur Texteinbettung und dem Amazon Neptune Medical Knowledge Graph interagiert.
-
Die Lambda-Funktion ist in den SageMaker AI-Texteinbettungsendpunkt integriert, um eine semantische Suche innerhalb der OpenCypher-Abfrage durchzuführen. Sie konvertiert die Abfrage in natürlicher Sprache in eine OpenCypher-Abfrage, indem sie die zugrunde liegende LangChain Agenten.
-
Die Lambda-Funktion fragt den Neptune Medical Knowledge Graph nach dem richtigen Datensatz ab und empfängt die Ausgabe aus dem Neptune Medical Knowledge Graph.
-
Die Lambda-Funktion gibt die Ergebnisse von Neptune an die Amazon Bedrock-Aktionsgruppen zurück.
-
Die Amazon Bedrock-Aktionsgruppen senden den abgerufenen Kontext an den Amazon Bedrock-Agenten.
-
Der Amazon Bedrock-Agent generiert die Antwort anhand der ursprünglichen Benutzerabfrage und des abgerufenen Kontextes aus dem Knowledge Graph.
LangChain Agenten für die Interaktion mit Diagrammen
Sie können integrieren LangChain mit Neptune, um graphbasierte Abfragen und Abrufe zu ermöglichen. Dieser Ansatz kann KI-gesteuerte Workflows verbessern, indem er die Graphdatenbankfunktionen von Neptune nutzt. Der Brauch LangChain Der Retriever fungiert als Vermittler. Das grundlegende Modell in Amazon Bedrock kann mit Neptune interagieren, indem es sowohl direkte Cypher-Abfragen als auch komplexere Graphalgorithmen verwendet.
Sie können den benutzerdefinierten Retriever verwenden, um zu verfeinern, wie LangChain Der Agent interagiert mit den Neptun-Graph-Algorithmen. Sie können beispielsweise Few-Shot-Prompting verwenden, um die Antworten des Basismodells auf der Grundlage bestimmter Muster oder Beispiele anzupassen. Sie können auch LLM-identifizierte Filter anwenden, um den Kontext zu verfeinern und die Genauigkeit der Antworten zu verbessern. Dies kann die Effizienz und Genauigkeit des gesamten Abrufprozesses bei der Interaktion mit komplexen Grafikdaten verbessern.
Die folgende Abbildung zeigt, wie ein benutzerdefinierter LangChain Der Agent orchestriert die Interaktion zwischen einem Amazon Bedrock Foundation-Modell und einem Amazon Neptune Medical Knowledge Graph.
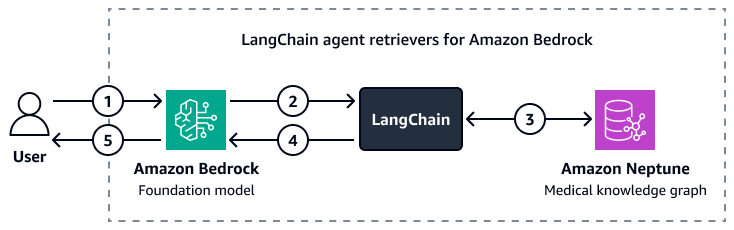
Das Diagramm zeigt den folgenden Workflow:
-
Ein Benutzer sendet eine Frage an Amazon Bedrock und LangChain Agent.
-
Das Amazon Bedrock Foundation-Modell verwendet das Neptun-Schema, das bereitgestellt wird von LangChain Agent, um eine Abfrage für die Frage des Benutzers zu generieren.
-
Das Tool LangChain Der Agent führt die Abfrage anhand des Amazon Neptune Medical Knowledge Graph aus.
-
Das Tool LangChain Der Agent sendet den abgerufenen Kontext an das Amazon Bedrock Foundation-Modell.
-
Das Amazon Bedrock Foundation-Modell verwendet den abgerufenen Kontext, um eine Antwort auf die Frage des Benutzers zu generieren.
Schritt 4: Erstellen einer Wissensdatenbank mit beschreibenden Echtzeitdaten
Als Nächstes erstellen Sie eine Wissensdatenbank mit beschreibenden Notizen zur Interaktion zwischen Arzt und Patient in Echtzeit, diagnostischen Bildbeurteilungen und Laboranalyseberichten. Bei dieser Wissensdatenbank handelt es sich um eine Vektordatenbank.
Nutzung einer medizinischen Wissensdatenbank von OpenSearch Service
Amazon OpenSearch Service kann große Mengen hochdimensionaler medizinischer Daten verwalten. Es handelt sich um einen verwalteten Service, der eine leistungsstarke Suche und Echtzeitanalysen ermöglicht. Es eignet sich gut als Vektordatenbank für RAG-Anwendungen. OpenSearch Der Service dient als Backend-Tool zur Verwaltung großer Mengen unstrukturierter oder halbstrukturierter Daten wie Krankenakten, Forschungsartikeln und klinischen Notizen. Seine fortschrittlichen semantischen Suchfunktionen helfen Ihnen dabei, kontextrelevante Informationen abzurufen. Dies macht es besonders nützlich in Anwendungen wie Systemen zur Unterstützung klinischer Entscheidungen, Tools zur Lösung von Patientenanfragen und Wissensmanagementsystemen im Gesundheitswesen. So kann ein Arzt beispielsweise schnell relevante Patientendaten oder Forschungsstudien finden, die bestimmten Symptomen oder Behandlungsprotokollen entsprechen. Dies hilft Ärzten dabei, Entscheidungen zu treffen, die sich auf die relevantesten up-to-date und relevantesten Informationen stützen.
OpenSearch Der Service kann skaliert und die Indizierung und Abfrage von Daten in Echtzeit abgewickelt werden. Dies macht es ideal für dynamische Umgebungen im Gesundheitswesen, in denen der zeitnahe Zugriff auf genaue Informationen entscheidend ist. Darüber hinaus verfügt es über multimodale Suchfunktionen, die sich optimal für Suchen eignen, die mehrere Eingaben erfordern, z. B. medizinische Bilder und Arztnotizen. Bei der Implementierung von OpenSearch Service for Healthcare-Anwendungen ist es wichtig, dass Sie präzise Felder und Zuordnungen definieren, um die Indexierung und den Datenabruf zu optimieren. Felder stellen die einzelnen Daten dar, z. B. Patientenakten, Krankengeschichten und Diagnosecodes. Zuordnungen definieren, wie diese Felder gespeichert (in eingebetteter Form oder Originalform) und abgefragt werden. Für Anwendungen im Gesundheitswesen ist es wichtig, Zuordnungen zu erstellen, die verschiedene Datentypen berücksichtigen, darunter strukturierte Daten (wie numerische Testergebnisse), halbstrukturierte Daten (wie Patientennotizen) und unstrukturierte Daten (wie medizinische Bilder)
In OpenSearch Service können Sie mithilfe kuratierter Eingabeaufforderungen neuronale Volltext-Suchanfragen
Erstellung einer RAG-Architektur
Sie können eine maßgeschneiderte RAG-Lösung bereitstellen, die Amazon Bedrock-Agenten verwendet, um eine medizinische Wissensdatenbank in OpenSearch Service abzufragen. Um dies zu erreichen, erstellen Sie eine AWS Lambda Funktion, die mit OpenSearch Service interagieren und Anfragen abfragen kann. Die Lambda-Funktion bettet die Eingabefrage des Benutzers ein, indem sie auf einen SageMaker AI-Texteinbettungsendpunkt zugreift. Der Amazon Bedrock-Agent übergibt zusätzliche Abfrageparameter als Eingaben an die Lambda-Funktion. Die Funktion fragt die medizinische Wissensdatenbank in OpenSearch Service ab, die den relevanten medizinischen Inhalt zurückgibt. Nachdem Sie die Lambda-Funktion eingerichtet haben, fügen Sie sie als Aktionsgruppe innerhalb des Amazon Bedrock-Agenten hinzu. Der Amazon Bedrock-Agent nimmt die Eingaben des Benutzers entgegen, identifiziert die erforderlichen Variablen, übergibt die Variablen und die Frage an die Lambda-Funktion und initiiert dann die Funktion. Die Funktion gibt einen Kontext zurück, der dem Foundation-Modell hilft, eine genauere Antwort auf die Frage des Benutzers zu geben.
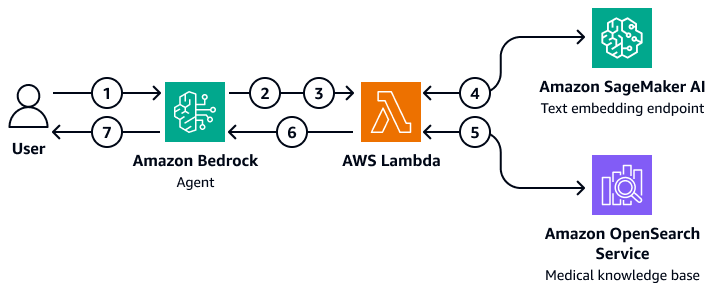
Das Diagramm zeigt den folgenden Workflow:
-
Ein Benutzer sendet eine Frage an den Amazon Bedrock-Agenten.
-
Der Amazon Bedrock-Agent wählt aus, welche Aktionsgruppe initiiert werden soll.
-
Der Amazon Bedrock-Agent initiiert eine AWS Lambda Funktion und übergibt ihr Parameter.
-
Die Lambda-Funktion initiiert das Amazon SageMaker AI-Texteinbettungsmodell, um die Benutzerfrage einzubetten.
-
Die Lambda-Funktion übergibt den eingebetteten Text und zusätzliche Parameter und Filter an Amazon OpenSearch Service. Amazon OpenSearch Service fragt die medizinische Wissensdatenbank ab und gibt die Ergebnisse an die Lambda-Funktion zurück.
-
Die Lambda-Funktion gibt die Ergebnisse an den Amazon Bedrock-Agenten zurück.
-
Das Basismodell im Amazon Bedrock-Agenten generiert eine Antwort auf der Grundlage der Ergebnisse und gibt die Antwort an den Benutzer zurück.
Für Situationen, in denen eine komplexere Filterung erforderlich ist, können Sie eine benutzerdefinierte Methode verwenden LangChain Retriever. Erstellen Sie diesen Retriever, indem Sie einen OpenSearch Service Vector Search Client einrichten, der direkt in LangChain. Diese Architektur ermöglicht es Ihnen, mehr Variablen zu übergeben, um die Filterparameter zu erstellen. Nachdem der Retriever eingerichtet ist, verwenden Sie das Amazon Bedrock-Modell und den Retriever, um eine Fragen-Antwort-Kette für den Abruf einzurichten. Diese Kette orchestriert die Interaktion zwischen dem Modell und dem Retriever, indem sie die Benutzereingaben und mögliche Filter an den Retriever weitergibt. Der Retriever gibt den relevanten Kontext zurück, der dem Foundation-Modell hilft, die Frage des Benutzers zu beantworten.
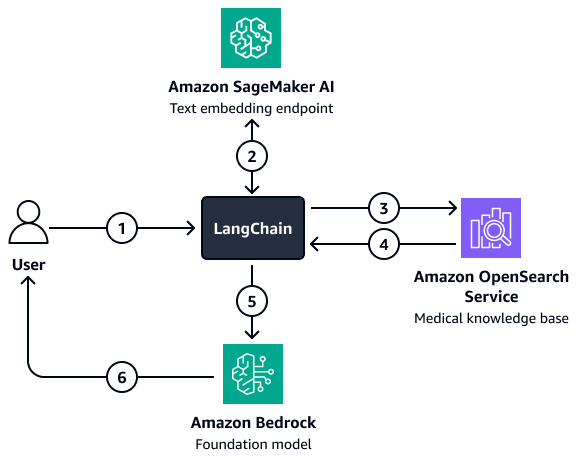
Das Diagramm zeigt den folgenden Workflow:
-
Ein Benutzer sendet eine Frage an den LangChain Retriever-Agent.
-
Das Tool LangChain Der Retriever-Agent sendet die Frage an den Amazon SageMaker AI-Texteinbettungsendpunkt, um die Frage einzubetten.
-
Das Tool LangChain Der Retriever-Agent leitet den eingebetteten Text an Amazon OpenSearch Service weiter.
-
Amazon OpenSearch Service sendet die abgerufenen Dokumente zurück an LangChain Retriever-Agent.
-
Das Tool LangChain Der Retriever-Agent leitet die Benutzerfrage und den abgerufenen Kontext an das Amazon Bedrock Foundation-Modell weiter.
-
Das Foundation-Modell generiert eine Antwort und sendet sie an den Benutzer.
Schritt 5: Verwendung LLMs zur Beantwortung medizinischer Fragen
Die vorherigen Schritte helfen Ihnen dabei, eine Anwendung für medizinische Intelligenz zu erstellen, mit der die Krankenakten eines Patienten abgerufen und relevante Medikamente und mögliche Diagnosen zusammengefasst werden können. Jetzt erstellen Sie die Generationsebene. Diese Ebene nutzt die generativen Funktionen eines LLM in Amazon Bedrock, wie Llama 3, um die Ausgabe der Anwendung zu verbessern.
Wenn ein Arzt eine Abfrage eingibt, führt die Kontext-Abruf-Ebene der Anwendung den Abrufvorgang aus dem Knowledge Graph durch und gibt die wichtigsten Datensätze zurück, die sich auf die Krankengeschichte, Demografie, Symptome, Diagnose und Behandlungsergebnisse des Patienten beziehen. Aus der Vektordatenbank werden außerdem in Echtzeit beschreibende Hinweise zur Interaktion zwischen Arzt und Patient, Erkenntnisse aus der diagnostischen Bildbeurteilung, Zusammenfassungen von Laboranalyseberichten und Erkenntnisse aus einer Vielzahl von medizinischen Forschungsarbeiten und wissenschaftlichen Büchern abgerufen. Diese am häufigsten abgerufenen Ergebnisse, die Anfrage des Klinikers und die Eingabeaufforderungen (die darauf zugeschnitten sind, Antworten auf der Grundlage der Art der Anfrage zu kuratieren) werden dann an das Foundation-Modell in Amazon Bedrock übergeben. Dies ist die Ebene zur Generierung von Antworten. Das LLM verwendet den abgerufenen Kontext, um eine Antwort auf die Anfrage des Klinikers zu generieren. Die folgende Abbildung zeigt den end-to-end Arbeitsablauf der Schritte in dieser Lösung.

Sie können ein vortrainiertes Basismodell in Amazon Bedrock, wie Llama 3, für eine Reihe von Anwendungsfällen verwenden, die die Anwendung für medizinische Intelligenz bewältigen muss. Das effektivste LLM für eine bestimmte Aufgabe hängt vom Anwendungsfall ab. Beispielsweise könnte ein vorab trainiertes Modell ausreichen, um Gespräche zwischen Patienten und Ärzten zusammenzufassen, Medikamente und Patientengeschichten zu durchsuchen und Erkenntnisse aus internen medizinischen Datensätzen und wissenschaftlichen Erkenntnissen abzurufen. Für andere komplexe Anwendungsfälle wie Laboruntersuchungen in Echtzeit, Empfehlungen für medizinische Verfahren und Prognosen von Behandlungsergebnissen könnte jedoch ein fein abgestimmtes LLM erforderlich sein. Sie können ein LLM verfeinern, indem Sie es anhand von Datensätzen aus dem medizinischen Bereich trainieren. Spezifische oder komplexe Anforderungen im Gesundheitswesen und in den Biowissenschaften treiben die Entwicklung dieser fein abgestimmten Modelle voran.
Weitere Informationen zur Feinabstimmung eines LLM oder zur Auswahl eines bestehenden LLM, das auf medizinischen Daten trainiert wurde, finden Sie unter Verwendung umfangreicher Sprachmodelle für Anwendungsfälle im Gesundheitswesen und in den Biowissenschaften.
Ausrichtung auf das AWS Well-Architected Framework
Die Lösung entspricht allen sechs Säulen des AWS Well-Architected Framework
-
Operative Exzellenz — Die Architektur ist entkoppelt, um eine effiziente Überwachung und Aktualisierung zu gewährleisten. Amazon Bedrock-Agenten AWS Lambda helfen Ihnen dabei, Tools schnell bereitzustellen und rückgängig zu machen.
-
Sicherheit — Diese Lösung wurde entwickelt, um Gesundheitsvorschriften wie HIPAA zu erfüllen. Sie können auch Verschlüsselung, detaillierte Zugriffskontrolle und Amazon Bedrock Guardrails implementieren, um Patientendaten zu schützen.
-
Zuverlässigkeit — AWS Managed Services wie Amazon OpenSearch Service und Amazon Bedrock bieten die Infrastruktur für eine kontinuierliche Modellinteraktion.
-
Leistungseffizienz — Die RAG-Lösung ruft mithilfe optimierter semantischer Suchen und Cypher-Abfragen schnell relevante Daten ab, während ein Agenten-Router optimale Routen für Benutzeranfragen identifiziert.
-
Kostenoptimierung — Das pay-per-token Modell in der Amazon Bedrock- und RAG-Architektur reduziert die Kosten für Inferenzen und Vorschulungen.
-
Nachhaltigkeit — Durch den Einsatz von serverloser Infrastruktur und pay-per-token Rechenleistung wird der Ressourcenverbrauch minimiert und die Nachhaltigkeit verbessert.