Die vorliegende Übersetzung wurde maschinell erstellt. Im Falle eines Konflikts oder eines Widerspruchs zwischen dieser übersetzten Fassung und der englischen Fassung (einschließlich infolge von Verzögerungen bei der Übersetzung) ist die englische Fassung maßgeblich.
Verwenden Sie SageMaker Processing für verteiltes Feature-Engineering von ML-Datensätzen im Terabyte-Bereich
Chris Boomhower, Amazon Web Services
Übersicht
Viele Datensätze im Terabyte-Bereich oder größer bestehen oft aus einer hierarchischen Ordnerstruktur, und die Dateien im Datensatz weisen manchmal wechselseitige Abhängigkeiten auf. Aus diesem Grund müssen Ingenieure und Datenwissenschaftler für maschinelles Lernen (ML) durchdachte Designentscheidungen treffen, um solche Daten für Modelltraining und Inferenz aufzubereiten. Dieses Muster zeigt, wie Sie manuelle Macrosharding- und Microsharding-Techniken in Kombination mit Amazon SageMaker Processing und virtueller CPU (vCPU) -Parallelisierung verwenden können, um Feature-Engineering-Prozesse für komplizierte Big-Data-ML-Datensätze effizient zu skalieren.
Dieses Muster definiert Macrosharding als die Aufteilung von Datenverzeichnissen auf mehrere Computer zur Verarbeitung und Microsharding als die Aufteilung von Daten auf jedem Computer über mehrere Verarbeitungsthreads. Das Muster demonstriert diese Techniken, indem Amazon SageMaker mit Beispiel-Zeitreihen-Wellenformaufzeichnungen aus dem MIMIC-III-Datensatz verwendet wirdPhysioNet .
Voraussetzungen und Einschränkungen
Voraussetzungen
Zugriff auf SageMaker Notebook-Instances oder SageMaker Studio, wenn Sie dieses Muster für Ihren eigenen Datensatz implementieren möchten. Wenn Sie Amazon SageMaker zum ersten Mal verwenden, finden Sie weitere Informationen unter Erste Schritte mit Amazon SageMaker in der AWS-Dokumentation.
SageMaker Studio, wenn Sie dieses Muster mit den PhysioNet MIMIC-III-Beispieldaten
implementieren möchten. Das Muster verwendet SageMaker Processing, erfordert jedoch keine Erfahrung mit der Ausführung von SageMaker Processing-Jobs.
Einschränkungen
Dieses Muster eignet sich gut für ML-Datensätze, die voneinander abhängige Dateien enthalten. Diese Interdependenzen profitieren am meisten von manuellem Macrosharding und der parallel Ausführung mehrerer SageMaker Einzelinstanz-Verarbeitungsjobs. Für Datensätze, bei denen solche Interdependenzen nicht existieren, ist die
ShardedByS3KeyFunktion in SageMaker Processing möglicherweise eine bessere Alternative zum Macrosharding, da sie fragmentierte Daten an mehrere Instanzen sendet, die von demselben Verarbeitungsauftrag verwaltet werden. Sie können jedoch die Microsharding-Strategie dieses Musters in beiden Szenarien implementieren, um Instanz v optimal zu nutzen. CPUs
Produktversionen
Amazon SageMaker Python SDK, Version 2
Architektur
Zieltechnologie-Stack
Amazon Simple Storage Service (Amazon-S3)
Amazon SageMaker
Zielarchitektur
Macrosharding und verteilte Instanzen EC2
Die 10 parallel Prozesse, die in dieser Architektur dargestellt werden, spiegeln die Struktur des MIMIC-III-Datensatzes wider. (Prozesse werden zur Vereinfachung des Diagramms durch Ellipsen dargestellt.) Eine ähnliche Architektur gilt für jeden Datensatz, wenn Sie manuelles Macrosharding verwenden. Im Fall von MIMIC-III können Sie die Rohstruktur des Datensatzes zu Ihrem Vorteil nutzen, indem Sie jeden Patientengruppenordner mit minimalem Aufwand separat verarbeiten. In der folgenden Abbildung wird der Block Datensatzgruppen auf der linken Seite angezeigt (1). Da die Daten verteilt sind, ist es sinnvoll, sie nach Patientengruppen aufzuteilen.
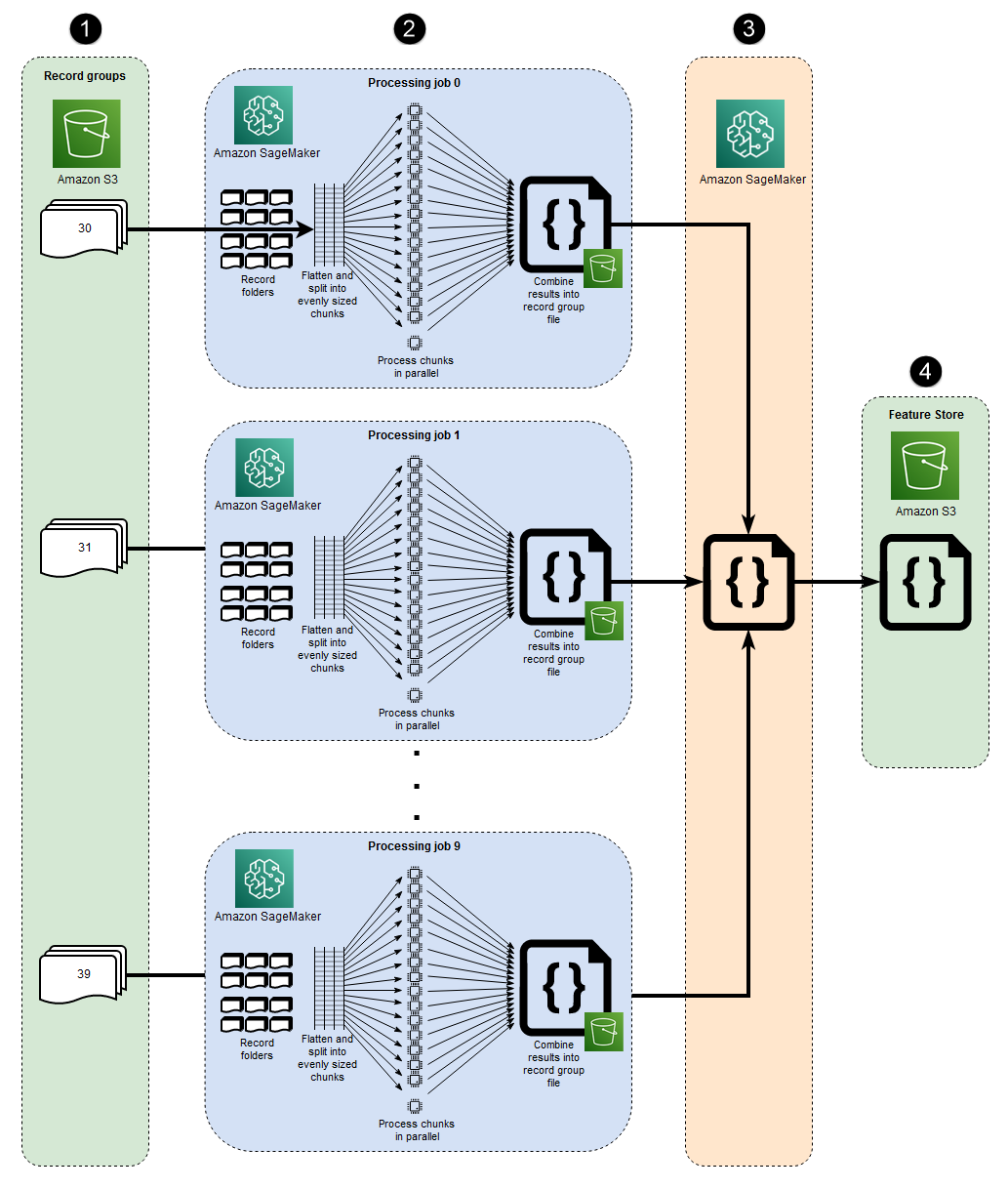
Manuelles Sharding nach Patientengruppen bedeutet jedoch, dass für jeden Patientengruppenordner ein separater Verarbeitungsauftrag erforderlich ist, wie Sie im mittleren Abschnitt des Diagramms (2) sehen können, und nicht ein einziger Verarbeitungsauftrag mit mehreren Instanzen. EC2 Da die Daten von MIMIC-III sowohl binäre Wellenformdateien als auch passende textbasierte Header-Dateien enthalten und für die binäre Datenextraktion eine Abhängigkeit von der WFDB-Bibliotheks3_data_distribution_type='FullyReplicated' wann Sie die Eingabe für den Verarbeitungsjob definieren. Wenn alle Daten in einem einzigen Verzeichnis verfügbar wären und es keine Abhängigkeiten zwischen den Dateien gäbe, könnte es alternativ besser sein, einen einzelnen Verarbeitungsauftrag mit mehreren EC2 Instanzen zu starten und anzugeben. s3_data_distribution_type='ShardedByS3Key' Wenn Sie ShardedByS3Key als Amazon S3 S3-Datenverteilungstyp angeben, wird SageMaker das Daten-Sharding automatisch über Instances hinweg verwaltet.
Das Starten eines Verarbeitungsauftrags für jeden Ordner ist eine kostengünstige Methode zur Vorverarbeitung der Daten, da die gleichzeitige Ausführung mehrerer Instances Zeit spart. Um zusätzliche Kosten und Zeit zu sparen, können Sie Microsharding für jeden Verarbeitungsauftrag verwenden.
Microsharding und paralleles V CPUs
Innerhalb jedes Verarbeitungsauftrags werden die gruppierten Daten weiter aufgeteilt, um die Nutzung aller verfügbaren V CPUs auf der SageMaker vollständig verwalteten EC2 Instanz zu maximieren. Die Blöcke im mittleren Abschnitt des Diagramms (2) zeigen, was innerhalb der einzelnen primären Verarbeitungsaufträge passiert. Der Inhalt der Patientenaktenordner wird vereinfacht und gleichmäßig auf der Grundlage der Anzahl der verfügbaren V CPUs auf der Instanz aufgeteilt. Nachdem der Ordnerinhalt aufgeteilt wurde, wird der Satz von Dateien mit gleichmäßiger Größe CPUs zur Verarbeitung auf alle V verteilt. Wenn die Verarbeitung abgeschlossen ist, werden die Ergebnisse jeder vCPU in einer einzigen Datendatei für jeden Verarbeitungsauftrag zusammengefasst.
Im beigefügten Code werden diese Konzepte im folgenden Abschnitt der src/feature-engineering-pass1/preprocessing.py Datei dargestellt.
def chunks(lst, n): """ Yield successive n-sized chunks from lst. :param lst: list of elements to be divided :param n: number of elements per chunk :type lst: list :type n: int :return: generator comprising evenly sized chunks :rtype: class 'generator' """ for i in range(0, len(lst), n): yield lst[i:i + n] # Generate list of data files on machine data_dir = input_dir d_subs = next(os.walk(os.path.join(data_dir, '.')))[1] file_list = [] for ds in d_subs: file_list.extend(os.listdir(os.path.join(data_dir, ds, '.'))) dat_list = [os.path.join(re.split('_|\.', f)[0].replace('n', ''), f[:-4]) for f in file_list if f[-4:] == '.dat'] # Split list of files into sub-lists cpu_count = multiprocessing.cpu_count() splits = int(len(dat_list) / cpu_count) if splits == 0: splits = 1 dat_chunks = list(chunks(dat_list, splits)) # Parallelize processing of sub-lists across CPUs ws_df_list = Parallel(n_jobs=-1, verbose=0)(delayed(run_process)(dc) for dc in dat_chunks) # Compile and pickle patient group dataframe ws_df_group = pd.concat(ws_df_list) ws_df_group = ws_df_group.reset_index().rename(columns={'index': 'signal'}) ws_df_group.to_json(os.path.join(output_dir, group_data_out))
Eine Funktion,chunks, wird zunächst so definiert, dass sie eine bestimmte Liste verarbeitet, indem sie sie in gleich große Längenabschnitte aufteilt n und diese Ergebnisse als Generator zurückgibt. Als Nächstes werden die Daten auf alle Patientenordner reduziert, indem eine Liste aller vorhandenen binären Wellenformdateien erstellt wird. Danach wird die Anzahl von v ermittelt, die auf der Instanz CPUs verfügbar ist. EC2 Die Liste der binären Wellenformdateien wird CPUs durch Aufruf gleichmäßig auf diese v aufgeteiltchunks, und dann wird jede Wellenform-Unterliste auf ihrer eigenen vCPU verarbeitet, indem die Parallel-Klasse von joblib verwendet wird.
Wenn alle anfänglichen Verarbeitungsaufträge abgeschlossen sind, kombiniert ein sekundärer Verarbeitungsauftrag, der im Block rechts neben dem Diagramm (3) dargestellt ist, die von jedem primären Verarbeitungsauftrag erstellten Ausgabedateien und schreibt die kombinierte Ausgabe in Amazon S3 (4).
Tools
Tools
Python
— Der für dieses Muster verwendete Beispielcode ist Python (Version 3). SageMaker Studio — Amazon SageMaker Studio ist eine webbasierte, integrierte Entwicklungsumgebung (IDE) für maschinelles Lernen, mit der Sie Ihre Machine-Learning-Modelle erstellen, trainieren, debuggen, bereitstellen und überwachen können. Sie führen SageMaker Verarbeitungsaufträge mithilfe von Jupyter-Notebooks in Studio aus. SageMaker
SageMaker Verarbeitung — Amazon SageMaker Processing bietet eine vereinfachte Möglichkeit, Ihre Datenverarbeitungs-Workloads auszuführen. In diesem Muster wird der Feature-Engineering-Code mithilfe von SageMaker Verarbeitungsaufträgen maßstabsgetreu implementiert.
Code
Die angehängte ZIP-Datei enthält den vollständigen Code für dieses Muster. Im folgenden Abschnitt werden die Schritte zum Erstellen der Architektur für dieses Muster beschrieben. Jeder Schritt wird durch einen Beispielcode aus dem Anhang veranschaulicht.
Epen
| Aufgabe | Beschreibung | Erforderliche Fähigkeiten |
|---|---|---|
| Greifen Sie auf Amazon SageMaker Studio zu. | Starten Sie SageMaker Studio über Ihr AWS-Konto, indem Sie den Anweisungen in der SageMaker Amazon-Dokumentation folgen. | Datenwissenschaftler, ML-Ingenieur |
| Installieren Sie das Hilfsprogramm wget. | Installieren Sie wget, wenn Sie mit einer neuen SageMaker Studio-Konfiguration vertraut sind oder wenn Sie diese Dienstprogramme noch nie in Studio verwendet haben. SageMaker Öffnen Sie zur Installation ein Terminalfenster in der SageMaker Studio-Konsole und führen Sie den folgenden Befehl aus:
| Datenwissenschaftler, ML-Ingenieur |
| Laden Sie den Beispielcode herunter und entpacken Sie ihn. | Laden Sie die
Navigieren Sie zu dem Ordner, in den Sie die ZIP-Datei extrahiert haben, und extrahieren Sie den Inhalt der
| Datenwissenschaftler, ML-Ingenieur |
| Laden Sie den Beispieldatensatz von physionet.org herunter und laden Sie ihn auf Amazon S3 hoch. | Führen Sie das | Datenwissenschaftler, ML-Ingenieur |
| Aufgabe | Beschreibung | Erforderliche Fähigkeiten |
|---|---|---|
| Reduzieren Sie die Dateihierarchie in allen Unterverzeichnissen. | In großen Datensätzen wie MIMIC-III sind Dateien häufig auf mehrere Unterverzeichnisse verteilt, selbst innerhalb einer logischen übergeordneten Gruppe. Ihr Skript sollte so konfiguriert sein, dass alle Gruppendateien in allen Unterverzeichnissen reduziert werden, wie der folgende Code zeigt.
Anmerkung Die Beispielcodefragmente in diesem Epos stammen aus der | Datenwissenschaftler, ML-Ingenieur |
| Teilen Sie Dateien basierend auf der Anzahl der vCPU in Untergruppen auf. | Dateien sollten in gleichmäßig große Untergruppen oder Blöcke aufgeteilt werden, abhängig von der Anzahl der V, die auf der Instanz CPUs vorhanden sind, die das Skript ausführt. Für diesen Schritt können Sie Code implementieren, der dem folgenden ähnelt.
| Datenwissenschaftler, ML-Ingenieur |
| Parallelisieren Sie die Verarbeitung von Untergruppen über v. CPUs | Die Skriptlogik sollte so konfiguriert sein, dass alle Untergruppen parallel verarbeitet werden. Verwenden Sie dazu die
| Datenwissenschaftler, ML-Ingenieur |
| Speichern Sie die Ausgabe einer einzelnen Dateigruppe in Amazon S3. | Wenn die parallel vCPU-Verarbeitung abgeschlossen ist, sollten die Ergebnisse jeder vCPU kombiniert und in den S3-Bucket-Pfad der Dateigruppe hochgeladen werden. Für diesen Schritt können Sie Code verwenden, der dem folgenden ähnelt.
| Datenwissenschaftler, ML-Ingenieur |
| Aufgabe | Beschreibung | Erforderliche Fähigkeiten |
|---|---|---|
| Kombinieren Sie Datendateien, die bei allen Verarbeitungsaufträgen erstellt wurden, bei denen das erste Skript ausgeführt wurde. | Das vorherige Skript gibt für jeden SageMaker Verarbeitungsjob, der eine Gruppe von Dateien aus dem Datensatz verarbeitet, eine einzelne Datei aus. Als Nächstes müssen Sie diese Ausgabedateien zu einem einzigen Objekt kombinieren und einen einzigen Ausgabedatensatz in Amazon S3 schreiben. Dies wird in der
| Datenwissenschaftler, ML-Ingenieur |
| Aufgabe | Beschreibung | Erforderliche Fähigkeiten |
|---|---|---|
| Führen Sie den ersten Verarbeitungsauftrag aus. | Um Macrosharding durchzuführen, führen Sie für jede Dateigruppe einen separaten Verarbeitungsauftrag aus. Microsharding wird innerhalb jedes Verarbeitungsauftrags ausgeführt, da jeder Job Ihr erstes Skript ausführt. Der folgende Code zeigt, wie ein Verarbeitungsauftrag für jedes Dateigruppenverzeichnis im folgenden Codeausschnitt (enthalten in) gestartet wird.
| Datenwissenschaftler, ML-Ingenieur |
| Führen Sie den zweiten Verarbeitungsauftrag aus. | Um die vom ersten Satz von Verarbeitungsaufträgen generierten Ausgaben zu kombinieren und zusätzliche Berechnungen für die Vorverarbeitung durchzuführen, führen Sie Ihr zweites Skript mit einem einzigen SageMaker Verarbeitungsauftrag aus. Der folgende Code veranschaulicht dies (enthalten in
| Datenwissenschaftler, ML-Ingenieur |
Zugehörige Ressourcen
Mit Quick Start in Amazon SageMaker Studio einsteigen (SageMaker Dokumentation)
Daten verarbeiten (SageMaker Dokumentation)
Datenverarbeitung mit Scikit-Learn (Dokumentation) SageMaker
Moody, B., Moody, G., Villarroel, M., Clifford, G.D., & Silva, I. (2020). MIMIC-III-Wellenform-Datenbank
(Version 1.0). PhysioNet. Johnson, A.E.W., Pollard, T.J., Shen, L., Lehman, L.H., Feng, M., Ghassemi, M., Moody, B., Szolovits, P., Celi, L.A., & Mark, R.G. (2016). MIMIC-III
, eine frei zugängliche Datenbank für Intensivpflege. Wissenschaftliche Daten, 3, 160035.