Las traducciones son generadas a través de traducción automática. En caso de conflicto entre la traducción y la version original de inglés, prevalecerá la version en inglés.
Utilice el SageMaker procesamiento para la ingeniería de características distribuidas de conjuntos de datos de aprendizaje automático a escala de terabytes
Chris Boomhower, Amazon Web Services
Resumen
Muchos conjuntos de datos a escala de terabytes o más grandes suelen constar de una estructura jerárquica de carpetas y, en ocasiones, los archivos del conjunto de datos comparten interdependencias. Por este motivo, los ingenieros de machine learning (ML) y los científicos de datos deben tomar decisiones de diseño bien pensadas a fin de preparar dichos datos para el entrenamiento y la inferencia de modelos. Este patrón demuestra cómo puede utilizar técnicas manuales de macrofragmentación y microfragmentación en combinación con Amazon SageMaker Processing y la paralelización de CPU virtual (vCPU) para escalar de manera eficiente los procesos de ingeniería de características para conjuntos de datos de aprendizaje automático de big data complicados.
Este patrón define la macrofragmentación como la división de los directorios de datos en varias máquinas para su procesamiento, y la microfragmentación como la división de los datos de cada máquina en varios subprocesos de procesamiento. El patrón demuestra estas técnicas mediante el uso de Amazon SageMaker con ejemplos de registros de formas de onda de series temporales del conjunto de datos PhysioNet MIMIC-III
Requisitos previos y limitaciones
Requisitos previos
Acceda a instancias de SageMaker notebook o a SageMaker Studio, si desea implementar este patrón en su propio conjunto de datos. Si es la primera vez que utiliza Amazon SageMaker , consulte Comenzar con Amazon SageMaker en la documentación de AWS.
SageMaker Studio, si quiere implementar este patrón con los datos de muestra del PhysioNet MIMIC-III
. El patrón usa SageMaker Processing, pero no requiere experiencia en la ejecución SageMaker de trabajos de Processing.
Limitaciones
Este patrón se adapta bien a los conjuntos de datos de machine learning que incluyen archivos interdependientes. Estas interdependencias son las que más se benefician de la fragmentación manual de macros y de la ejecución en paralelo de varios trabajos de procesamiento de una sola instancia SageMaker . Para los conjuntos de datos en los que no existen dichas interdependencias, la
ShardedByS3Keyfunción de SageMaker procesamiento podría ser una mejor alternativa a la macrofragmentación, ya que envía los datos fragmentados a varias instancias administradas por el mismo trabajo de procesamiento. Sin embargo, puede implementar la estrategia de microfragmentación de este patrón en ambos escenarios para aprovechar al máximo la instancia v. CPUs
Versiones de producto
Amazon SageMaker Python SDK versión 2
Arquitectura
Pila de tecnología de destino
Amazon Simple Storage Service (Amazon S3)
Amazon SageMaker
Arquitectura de destino
Macrofragmentación e instancias distribuidas EC2
Los 10 procesos paralelos representados en esta arquitectura reflejan la estructura del conjunto de datos MIMIC-III. (Los procesos se representan mediante elipses para simplificar el diagrama). Cuando se utiliza la macrofragmentación manual, se aplica una arquitectura similar a cualquier conjunto de datos. En el caso de MIMIC-III, puede aprovechar la estructura sin procesar del conjunto de datos procesando la carpeta de cada grupo de pacientes por separado, con un esfuerzo mínimo. En el siguiente diagrama, el bloque de grupos de registros aparece a la izquierda (1). Dada la naturaleza distribuida de los datos, tiene sentido dividirlos por grupo de pacientes.
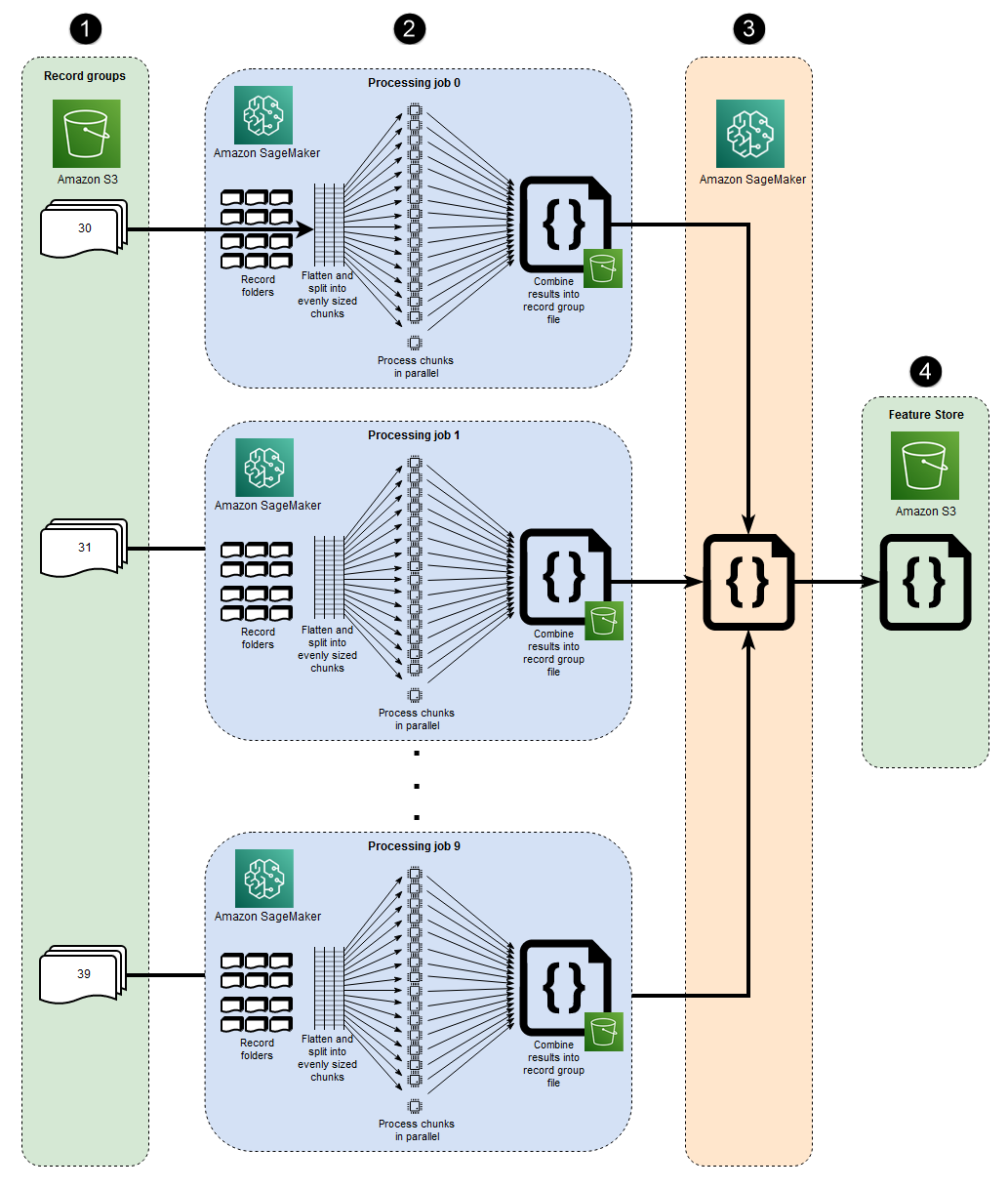
Sin embargo, la fragmentación manual por grupo de pacientes significa que se requiere un trabajo de procesamiento independiente para cada carpeta del grupo de pacientes, como puede ver en la sección central del diagrama (2), en lugar de un solo trabajo de procesamiento con varias instancias. EC2 Como los datos de MIMIC-III incluyen tanto archivos de forma de onda binarios como archivos de encabezados basados en texto coincidentes, y existe una dependencia obligatoria de la biblioteca wfdbs3_data_distribution_type='FullyReplicated' cuándo se define la entrada del trabajo de procesamiento. Como alternativa, si todos los datos estuvieran disponibles en un único directorio y no existieran dependencias entre los archivos, una opción más adecuada sería lanzar un único trabajo de procesamiento con varias EC2 instancias especificadass3_data_distribution_type='ShardedByS3Key'. Si ShardedByS3Key se especifica el tipo de distribución de datos de Amazon S3, se SageMaker gestionará automáticamente la fragmentación de datos en todas las instancias.
Lanzar un trabajo de procesamiento para cada carpeta es una forma rentable de preprocesar los datos, ya que la ejecución simultánea de varias instancias ahorra tiempo. Para ahorrar costos y tiempo adicionales, puede utilizar la microfragmentación en cada trabajo de procesamiento.
Microsharding y parallel v CPUs
Dentro de cada trabajo de procesamiento, los datos agrupados se dividen aún más para maximizar el uso de todos los v disponibles CPUs en la instancia SageMaker totalmente administrada EC2 . Los bloques de la sección central del diagrama (2) muestran lo que ocurre en cada trabajo de procesamiento principal. El contenido de las carpetas de registros de pacientes se aplana y se divide en partes iguales en función del número de v disponibles CPUs en la instancia. Una vez dividido el contenido de la carpeta, el conjunto de archivos de tamaño uniforme se distribuye por todos los archivos v CPUs para su procesamiento. Cuando se completa el procesamiento, los resultados de cada vCPU se combinan en un único archivo de datos para cada trabajo de procesamiento.
En el código adjunto, estos conceptos se representan en la siguiente sección del archivo src/feature-engineering-pass1/preprocessing.py.
def chunks(lst, n): """ Yield successive n-sized chunks from lst. :param lst: list of elements to be divided :param n: number of elements per chunk :type lst: list :type n: int :return: generator comprising evenly sized chunks :rtype: class 'generator' """ for i in range(0, len(lst), n): yield lst[i:i + n] # Generate list of data files on machine data_dir = input_dir d_subs = next(os.walk(os.path.join(data_dir, '.')))[1] file_list = [] for ds in d_subs: file_list.extend(os.listdir(os.path.join(data_dir, ds, '.'))) dat_list = [os.path.join(re.split('_|\.', f)[0].replace('n', ''), f[:-4]) for f in file_list if f[-4:] == '.dat'] # Split list of files into sub-lists cpu_count = multiprocessing.cpu_count() splits = int(len(dat_list) / cpu_count) if splits == 0: splits = 1 dat_chunks = list(chunks(dat_list, splits)) # Parallelize processing of sub-lists across CPUs ws_df_list = Parallel(n_jobs=-1, verbose=0)(delayed(run_process)(dc) for dc in dat_chunks) # Compile and pickle patient group dataframe ws_df_group = pd.concat(ws_df_list) ws_df_group = ws_df_group.reset_index().rename(columns={'index': 'signal'}) ws_df_group.to_json(os.path.join(output_dir, group_data_out))
Una función, chunks, se define primero para consumir una lista dada dividiéndola en trozos de longitud de tamaño uniforme n y devolviendo estos resultados como un generador. A continuación, los datos se agrupan en las carpetas de los pacientes mediante la compilación de una lista de todos los archivos de forma de onda binaria presentes. Una vez hecho esto, se obtiene el número de v CPUs disponible en la EC2 instancia. La lista de archivos de formas de onda binarias se divide uniformemente entre estas v CPUs mediante una llamada ychunks, a continuación, cada sublista de formas de onda se procesa en su propia vCPU mediante la clase Parallel de joblib.
Cuando se hayan completado todos los trabajos de procesamiento iniciales, un trabajo de procesamiento secundario, que se muestra en el bloque a la derecha del diagrama (3), combina los archivos de salida generados por cada trabajo de procesamiento principal y escribe el resultado combinado en Amazon S3 (4).
Herramientas
Herramientas
Python
: el código de ejemplo utilizado para este patrón es Python (versión 3). SageMaker Studio: Amazon SageMaker Studio es un entorno de desarrollo integrado (IDE) basado en la web para el aprendizaje automático que le permite crear, entrenar, depurar, implementar y supervisar sus modelos de aprendizaje automático. Los trabajos de SageMaker procesamiento se ejecutan con los cuadernos de Jupyter incluidos en Studio. SageMaker
SageMaker Procesamiento: Amazon SageMaker Processing proporciona una forma simplificada de ejecutar sus cargas de trabajo de procesamiento de datos. En este patrón, el código de ingeniería de funciones se implementa a escala mediante tareas SageMaker de procesamiento.
Código
El archivo .zip adjunto proporciona el código completo de este patrón. En la siguiente sección se describen los pasos para crear la arquitectura para este patrón. Cada paso se ilustra con un ejemplo de código del archivo adjunto.
Epics
| Tarea | Descripción | Habilidades requeridas |
|---|---|---|
| Accede a Amazon SageMaker Studio. | Inscríbase en SageMaker Studio en su cuenta de AWS siguiendo las instrucciones que se proporcionan en la SageMaker documentación de Amazon. | Científico de datos, ingeniero de machine learning |
| Instale la utilidad wget. | Instala wget si has incorporado una nueva configuración de SageMaker Studio o si nunca has utilizado estas utilidades en SageMaker Studio. Para instalarlo, abre una ventana de terminal en la consola de SageMaker Studio y ejecuta el siguiente comando:
| Científico de datos, ingeniero de machine learning |
| Descargue y descomprima el código de muestra. | Descargue el archivo
Desplácese hasta la ubicación donde descargó el archivo .zip y extraiga el contenido del archivo
| Científico de datos, ingeniero de machine learning |
| Descargue el conjunto de datos de muestra de physionet.org y cárguelo en Amazon S3. | Ejecute el cuaderno de Jupyter | Científico de datos, ingeniero de machine learning |
| Tarea | Descripción | Habilidades requeridas |
|---|---|---|
| Aplane la jerarquía de archivos en todos los subdirectorios. | En conjuntos de datos grandes, como MIMIC-III, los archivos suelen distribuirse en varios subdirectorios, incluso dentro de un grupo principal lógico. El script debe estar configurado para aplanar todos los archivos del grupo en todos los subdirectorios, como se muestra en el siguiente código.
nota Los fragmentos de código de ejemplo de esta epopeya provienen del archivo que se proporciona en el | Científico de datos, ingeniero de machine learning |
| Divida los archivos en subgrupos según el recuento de vCPU. | Los archivos deben dividirse en subgrupos o fragmentos de tamaño uniforme, según el número de v CPUs presentes en la instancia que ejecuta el script. Para este paso, puede implementar código similar al siguiente.
| Científico de datos, ingeniero de machine learning |
| Paralelice el procesamiento de los subgrupos entre v. CPUs | La lógica del script debe configurarse para procesar todos los subgrupos en paralelo. Para ello, utilice la clase
| Científico de datos, ingeniero de machine learning |
| Guarde la salida de un solo grupo de archivos en Amazon S3. | Cuando se complete el procesamiento de la vCPU paralela, los resultados de cada vCPU deben combinarse y cargarse en la ruta del bucket de S3 del grupo de archivos. Para este paso, puede utilizar código similar al siguiente.
| Científico de datos, ingeniero de machine learning |
| Tarea | Descripción | Habilidades requeridas |
|---|---|---|
| Combine los archivos de datos generados en todos los trabajos de procesamiento en los que se ejecutó el primer script. | El script anterior genera un único archivo para cada trabajo de SageMaker procesamiento que procesa un grupo de archivos del conjunto de datos. A continuación, debe combinar estos archivos de salida en un único objeto y escribir un único conjunto de datos de salida en Amazon S3. Esto se demuestra en el archivo
| Científico de datos, ingeniero de machine learning |
| Tarea | Descripción | Habilidades requeridas |
|---|---|---|
| Ejecute el primer trabajo de procesamiento. | Para realizar la fragmentación de macros, ejecute un trabajo de procesamiento independiente para cada grupo de archivos. La microfragmentación se realiza dentro de cada trabajo de procesamiento, ya que cada trabajo ejecuta el primer script. El código siguiente muestra cómo iniciar un trabajo de procesamiento para cada directorio de grupos de archivos en el siguiente fragmento (incluido en
| Científico de datos, ingeniero de machine learning |
| Ejecute el segundo trabajo de procesamiento. | Para combinar los resultados generados por el primer conjunto de trabajos de procesamiento y realizar cualquier cálculo adicional para el preprocesamiento, ejecute el segundo script mediante un único trabajo de SageMaker procesamiento. El siguiente código lo demuestra (incluido en
| Científico de datos, ingeniero de machine learning |
Recursos relacionados
Incorporarse a Amazon SageMaker Studio mediante Quick Start (SageMaker documentación)
Datos del proceso (SageMaker documentación)
Procesamiento de datos con scikit-learn (documentación) SageMaker
Moody, B., Moody, G., Villarroel, M., Clifford, G. D., & Silva, I. (2020). Base de datos de formas de onda MIMIC-III (versión 1.0)
. PhysioNet. Johnson, A. E. W., Pollard, T. J., Shen, L., Lehman, L. H., Feng, M., Ghassemi, M., Moody, B., Szolovits, P., Celi, L. A., & Mark, R. G. (2016). MIMIC-III, una base de datos de cuidados intensivos de acceso gratuito
. Scientific Data, 3, 160035.
Conexiones
Para acceder al contenido adicional asociado a este documento, descomprima el archivo: attachment.zip