Les traductions sont fournies par des outils de traduction automatique. En cas de conflit entre le contenu d'une traduction et celui de la version originale en anglais, la version anglaise prévaudra.
Utiliser SageMaker le traitement pour l'ingénierie des fonctionnalités distribuées d'ensembles de données ML à l'échelle du téraoctet
Chris Boomhower, Amazon Web Services
Récapitulatif
De nombreux ensembles de données de plusieurs téraoctets ou plus se composent souvent d'une structure de dossiers hiérarchique, et les fichiers du jeu de données partagent parfois des interdépendances. C'est pourquoi les ingénieurs en apprentissage automatique (ML) et les scientifiques des données doivent prendre des décisions de conception réfléchies afin de préparer ces données pour l'entraînement et l'inférence des modèles. Ce modèle montre comment vous pouvez utiliser des techniques manuelles de macrosharding et de microsharding en combinaison avec Amazon SageMaker Processing et la parallélisation des processeurs virtuels (vCPU) pour adapter efficacement les processus d'ingénierie des fonctionnalités aux ensembles de données Big Data ML complexes.
Ce modèle définit le macrosharding comme la division de répertoires de données sur plusieurs machines pour le traitement, et le microsharding comme le partage des données de chaque machine sur plusieurs threads de traitement. Le modèle illustre ces techniques en utilisant Amazon SageMaker avec des exemples d'enregistrements de formes d'onde de séries chronologiques issus du jeu de données PhysioNet MIMIC-III
Conditions préalables et limitations
Prérequis
Accès aux instances de SageMaker bloc-notes ou à SageMaker Studio, si vous souhaitez implémenter ce modèle pour votre propre ensemble de données. Si vous utilisez Amazon SageMaker pour la première fois, consultez la section Commencer avec Amazon SageMaker dans la documentation AWS.
SageMaker Studio, si vous souhaitez implémenter ce modèle avec les exemples de données PhysioNet MIMIC-III
. Le modèle utilise le SageMaker traitement, mais ne nécessite aucune expérience dans l'exécution de tâches SageMaker de traitement.
Limites
Ce modèle convient parfaitement aux ensembles de données ML qui incluent des fichiers interdépendants. Ces interdépendances tirent le meilleur parti du macrosharding manuel et de l'exécution en parallèle de plusieurs SageMaker tâches de traitement en instance unique. Pour les ensembles de données où de telles interdépendances n'existent pas, la
ShardedByS3Keyfonctionnalité de SageMaker Processing peut constituer une meilleure alternative au macrosharding, car elle envoie des données fragmentées à plusieurs instances gérées par la même tâche de traitement. Cependant, vous pouvez implémenter la stratégie de microsharding de ce modèle dans les deux scénarios afin d'utiliser au mieux l'instance v. CPUs
Versions du produit
Kit de développement logiciel Amazon SageMaker Python version 2
Architecture
Pile technologique cible
Amazon Simple Storage Service (Amazon S3)
Amazon SageMaker
Architecture cible
Macrosharding et instances distribuées EC2
Les 10 processus parallèles représentés dans cette architecture reflètent la structure du jeu de données MIMIC-III. (Les processus sont représentés par des ellipses pour simplifier les diagrammes.) Une architecture similaire s'applique à n'importe quel ensemble de données lorsque vous utilisez le macrosharding manuel. Dans le cas de MIMIC-III, vous pouvez utiliser la structure brute de l'ensemble de données à votre avantage en traitant chaque dossier de groupe de patients séparément, avec un minimum d'effort. Dans le schéma suivant, le bloc des groupes d'enregistrements apparaît sur la gauche (1). Compte tenu de la nature distribuée des données, il est logique de les partager par groupe de patients.
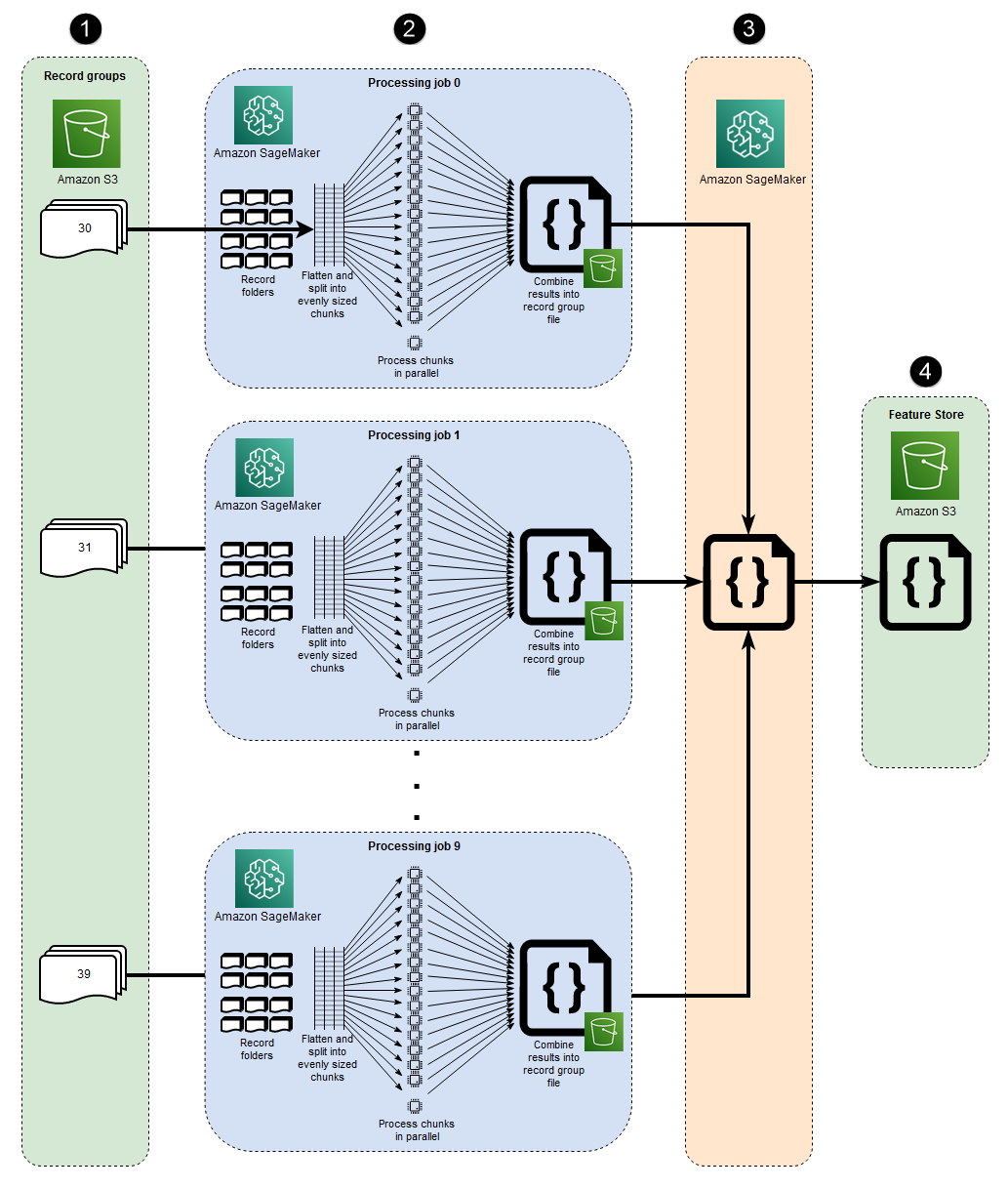
Toutefois, le découpage manuel par groupe de patients signifie qu'une tâche de traitement distincte est requise pour chaque dossier de groupe de patients, comme vous pouvez le voir dans la partie centrale du diagramme (2), au lieu d'une tâche de traitement unique avec plusieurs EC2 instances. Étant donné que les données de MIMIC-III incluent à la fois des fichiers de formes d'onde binaires et des fichiers d'en-tête textuels correspondants, et que l'extraction de données binaires nécessite une dépendance à la bibliothèque wfdbs3_data_distribution_type='FullyReplicated' quand vous définissez l'entrée de la tâche de traitement. Sinon, si toutes les données étaient disponibles dans un seul répertoire et qu'aucune dépendance n'existait entre les fichiers, une option plus appropriée pourrait être de lancer une seule tâche de traitement avec plusieurs EC2 instances s3_data_distribution_type='ShardedByS3Key' spécifiées. Spécifier ShardedByS3Key comme le type de distribution de données Amazon S3 indique SageMaker de gérer automatiquement le partitionnement des données entre les instances.
Le lancement d'une tâche de traitement pour chaque dossier est un moyen rentable de prétraiter les données, car l'exécution simultanée de plusieurs instances permet de gagner du temps. Pour économiser du temps et des coûts supplémentaires, vous pouvez utiliser le microsharding dans chaque tâche de traitement.
Microsharding et parallel v CPUs
Au sein de chaque tâche de traitement, les données groupées sont ensuite divisées afin de maximiser l'utilisation de tous les v disponibles CPUs sur l' EC2 instance SageMaker entièrement gérée. Les blocs situés dans la partie centrale du diagramme (2) décrivent ce qui se passe dans le cadre de chaque tâche de traitement principale. Le contenu des dossiers des patients est aplati et divisé de manière égale en fonction du nombre de v disponibles CPUs sur l'instance. Une fois le contenu du dossier divisé, l'ensemble de fichiers de taille uniforme est réparti sur tous les v CPUs pour être traité. Une fois le traitement terminé, les résultats de chaque vCPU sont combinés dans un seul fichier de données pour chaque tâche de traitement.
Dans le code joint, ces concepts sont représentés dans la section suivante du src/feature-engineering-pass1/preprocessing.py fichier.
def chunks(lst, n): """ Yield successive n-sized chunks from lst. :param lst: list of elements to be divided :param n: number of elements per chunk :type lst: list :type n: int :return: generator comprising evenly sized chunks :rtype: class 'generator' """ for i in range(0, len(lst), n): yield lst[i:i + n] # Generate list of data files on machine data_dir = input_dir d_subs = next(os.walk(os.path.join(data_dir, '.')))[1] file_list = [] for ds in d_subs: file_list.extend(os.listdir(os.path.join(data_dir, ds, '.'))) dat_list = [os.path.join(re.split('_|\.', f)[0].replace('n', ''), f[:-4]) for f in file_list if f[-4:] == '.dat'] # Split list of files into sub-lists cpu_count = multiprocessing.cpu_count() splits = int(len(dat_list) / cpu_count) if splits == 0: splits = 1 dat_chunks = list(chunks(dat_list, splits)) # Parallelize processing of sub-lists across CPUs ws_df_list = Parallel(n_jobs=-1, verbose=0)(delayed(run_process)(dc) for dc in dat_chunks) # Compile and pickle patient group dataframe ws_df_group = pd.concat(ws_df_list) ws_df_group = ws_df_group.reset_index().rename(columns={'index': 'signal'}) ws_df_group.to_json(os.path.join(output_dir, group_data_out))
Une fonction est d'abord définie pour consommer une liste donnée en la divisant en morceaux de taille égale n et en renvoyant ces résultats sous forme de générateur. chunks Ensuite, les données sont aplaties dans les dossiers des patients en compilant une liste de tous les fichiers de formes d'onde binaires présents. Une fois cela fait, le nombre de v CPUs disponibles sur l' EC2 instance est obtenu. La liste des fichiers de formes d'onde binaires est répartie uniformément sur ces v CPUs par appelchunks, puis chaque sous-liste de formes d'onde est traitée sur son propre vCPU en utilisant la classe Parallel de joblib.
Lorsque toutes les tâches de traitement initiales sont terminées, une tâche de traitement secondaire, illustrée dans le bloc à droite du diagramme (3), combine les fichiers de sortie produits par chaque tâche de traitement principale et écrit la sortie combinée sur Amazon S3 (4).
Outils
Outils
Python
— L'exemple de code utilisé pour ce modèle est Python (version 3). SageMaker Studio — Amazon SageMaker Studio est un environnement de développement intégré (IDE) basé sur le Web pour l'apprentissage automatique qui vous permet de créer, de former, de déboguer, de déployer et de surveiller vos modèles d'apprentissage automatique. Vous exécutez SageMaker des tâches de traitement en utilisant des blocs-notes Jupyter dans Studio. SageMaker
SageMaker Traitement — Amazon SageMaker Processing fournit un moyen simplifié d'exécuter vos charges de travail de traitement des données. Dans ce modèle, le code d'ingénierie des fonctionnalités est implémenté à grande échelle à l'aide de tâches SageMaker de traitement.
Code
Le fichier .zip joint fournit le code complet de ce modèle. La section suivante décrit les étapes à suivre pour créer l'architecture de ce modèle. Chaque étape est illustrée par un exemple de code extrait de la pièce jointe.
Épopées
| Tâche | Description | Compétences requises |
|---|---|---|
| Accédez à Amazon SageMaker Studio. | Accédez à SageMaker Studio depuis votre compte AWS en suivant les instructions fournies dans la SageMaker documentation Amazon. | Scientifique des données, ingénieur ML |
| Installez l'utilitaire wget. | Installez wget si vous avez intégré une nouvelle configuration de SageMaker Studio ou si vous n'avez jamais utilisé ces utilitaires dans SageMaker Studio auparavant. Pour l'installer, ouvrez une fenêtre de terminal dans la console SageMaker Studio et exécutez la commande suivante :
| Scientifique des données, ingénieur ML |
| Téléchargez et décompressez l'exemple de code. | Téléchargez le
Accédez au dossier dans lequel vous avez extrait le fichier .zip et extrayez le contenu du
| Scientifique des données, ingénieur ML |
| Téléchargez l'exemple de jeu de données sur physionet.org et chargez-le sur Amazon S3. | Exécutez le bloc-notes | Scientifique des données, ingénieur ML |
| Tâche | Description | Compétences requises |
|---|---|---|
| Aplatissez la hiérarchie des fichiers dans tous les sous-répertoires. | Dans les grands ensembles de données tels que MIMIC-III, les fichiers sont souvent répartis dans plusieurs sous-répertoires, même au sein d'un groupe parent logique. Votre script doit être configuré pour aplatir tous les fichiers de groupe dans tous les sous-répertoires, comme le montre le code suivant.
Note Les exemples d'extraits de code présentés dans cette épopée proviennent du | Scientifique des données, ingénieur ML |
| Divisez les fichiers en sous-groupes en fonction du nombre de vCPU. | Les fichiers doivent être divisés en sous-groupes ou en morceaux de taille égale, en fonction du nombre de v CPUs présents sur l'instance qui exécute le script. Pour cette étape, vous pouvez implémenter un code similaire au suivant.
| Scientifique des données, ingénieur ML |
| Paralléliser le traitement des sous-groupes dans v. CPUs | La logique du script doit être configurée pour traiter tous les sous-groupes en parallèle. Pour ce faire, utilisez la
| Scientifique des données, ingénieur ML |
| Enregistrez la sortie d'un seul groupe de fichiers sur Amazon S3. | Lorsque le traitement parallèle des vCPU est terminé, les résultats de chaque vCPU doivent être combinés et téléchargés dans le chemin du compartiment S3 du groupe de fichiers. Pour cette étape, vous pouvez utiliser un code similaire au suivant.
| Scientifique des données, ingénieur ML |
| Tâche | Description | Compétences requises |
|---|---|---|
| Combinez les fichiers de données produits dans toutes les tâches de traitement qui ont exécuté le premier script. | Le script précédent génère un fichier unique pour chaque tâche de SageMaker traitement qui traite un groupe de fichiers de l'ensemble de données. Ensuite, vous devez combiner ces fichiers de sortie en un seul objet et écrire un ensemble de données de sortie unique sur Amazon S3. Cela est démontré dans le
| Scientifique des données, ingénieur ML |
| Tâche | Description | Compétences requises |
|---|---|---|
| Exécutez la première tâche de traitement. | Pour effectuer le macrosharding, exécutez une tâche de traitement distincte pour chaque groupe de fichiers. Le microsharding est effectué dans chaque tâche de traitement, car chaque tâche exécute votre premier script. Le code suivant montre comment lancer une tâche de traitement pour chaque répertoire de groupe de fichiers dans l'extrait suivant (inclus dans
| Scientifique des données, ingénieur ML |
| Exécutez le deuxième travail de traitement. | Pour combiner les sorties générées par le premier ensemble de tâches de traitement et effectuer des calculs supplémentaires pour le prétraitement, vous devez exécuter votre deuxième script à l'aide d'une seule tâche de SageMaker traitement. Le code suivant illustre cela (inclus dans
| Scientifique des données, ingénieur ML |
Ressources connexes
Intégration à Amazon SageMaker Studio à l'aide de Quick Start (SageMaker documentation)
Données de processus (SageMaker documentation)
Traitement des données avec scikit-learn (documentation) SageMaker
Moody, B., Moody, G., Villarroel, M., Clifford, G.D., & Silva, I. (2020). Base de données de formes d'onde MIMIC-III
(version 1.0). PhysioNet. Johnson, A.E.W., Pollard, T.J., Shen, L., Lehman, L.H., Feng, M., Ghassemi, M., Moody, B., Szolovits, P., Celi, L.A., et Mark, R.G. (2016). MIMIC-III, une base de données sur les soins intensifs accessible gratuitement
. Données scientifiques, 3, 160035.