Le traduzioni sono generate tramite traduzione automatica. In caso di conflitto tra il contenuto di una traduzione e la versione originale in Inglese, quest'ultima prevarrà.
Usa SageMaker Processing per l'ingegneria di funzionalità distribuite di set di dati ML su scala terabyte
Chris Boomhower, Amazon Web Services
Riepilogo
Molti set di dati su scala terabyte o più grandi spesso sono costituiti da una struttura gerarchica di cartelle e i file del set di dati a volte condividono interdipendenze. Per questo motivo, gli ingegneri del machine learning (ML) e i data scientist devono prendere decisioni progettuali ponderate per preparare tali dati per l'addestramento e l'inferenza dei modelli. Questo modello dimostra come è possibile utilizzare tecniche manuali di macrosharding e microsharding in combinazione con Amazon SageMaker Processing e la parallelizzazione della CPU virtuale (vCPU) per scalare in modo efficiente i processi di progettazione delle funzionalità per complessi set di dati ML di big data.
Questo modello definisce il macrosharding come la suddivisione di directory di dati su più macchine per l'elaborazione e il microsharding come la suddivisione dei dati su ogni macchina su più thread di elaborazione. Il modello dimostra queste tecniche utilizzando Amazon SageMaker con esempi di record di forme d'onda di serie temporali dal set di dati MIMIC-III. PhysioNet
Prerequisiti e limitazioni
Prerequisiti
Accedi alle istanze di SageMaker notebook o a SageMaker Studio, se desideri implementare questo modello per il tuo set di dati. Se utilizzi Amazon SageMaker per la prima volta, consulta la sezione Guida introduttiva ad Amazon SageMaker nella documentazione AWS.
SageMaker Studio, se desideri implementare questo modello con i dati di esempio PhysioNet MIMIC-III
. Il pattern utilizza SageMaker Processing, ma non richiede alcuna esperienza nell'esecuzione SageMaker dei job di Processing.
Limitazioni
Questo modello è adatto ai set di dati ML che includono file interdipendenti. Queste interdipendenze traggono il massimo vantaggio dal macrosharding manuale e dall'esecuzione in parallelo di più processi di elaborazione a istanza singola SageMaker . Per i set di dati in cui tali interdipendenze non esistono, la
ShardedByS3Keyfunzionalità di SageMaker Processing potrebbe essere un'alternativa migliore al macrosharding, poiché invia dati suddivisi a più istanze gestite dallo stesso processo di elaborazione. Tuttavia, è possibile implementare la strategia di microsharding di questo pattern in entrambi gli scenari per utilizzare al meglio l'istanza v. CPUs
Versioni del prodotto
SDK Amazon SageMaker Python versione 2
Architettura
Stack tecnologico Target
Amazon Simple Storage Service (Amazon S3)
Amazon SageMaker
Architettura Target
Macrosharding e istanze distribuite EC2
I 10 processi paralleli rappresentati in questa architettura riflettono la struttura del set di dati MIMIC-III. (I processi sono rappresentati da ellissi per semplificare i diagrammi.) Un'architettura simile si applica a qualsiasi set di dati quando si utilizza il macrosharding manuale. Nel caso di MIMIC-III, è possibile utilizzare a proprio vantaggio la struttura grezza del set di dati elaborando ogni cartella del gruppo di pazienti separatamente, con il minimo sforzo. Nel diagramma seguente, il blocco dei gruppi di record appare sulla sinistra (1). Data la natura distribuita dei dati, ha senso condividerli per gruppo di pazienti.
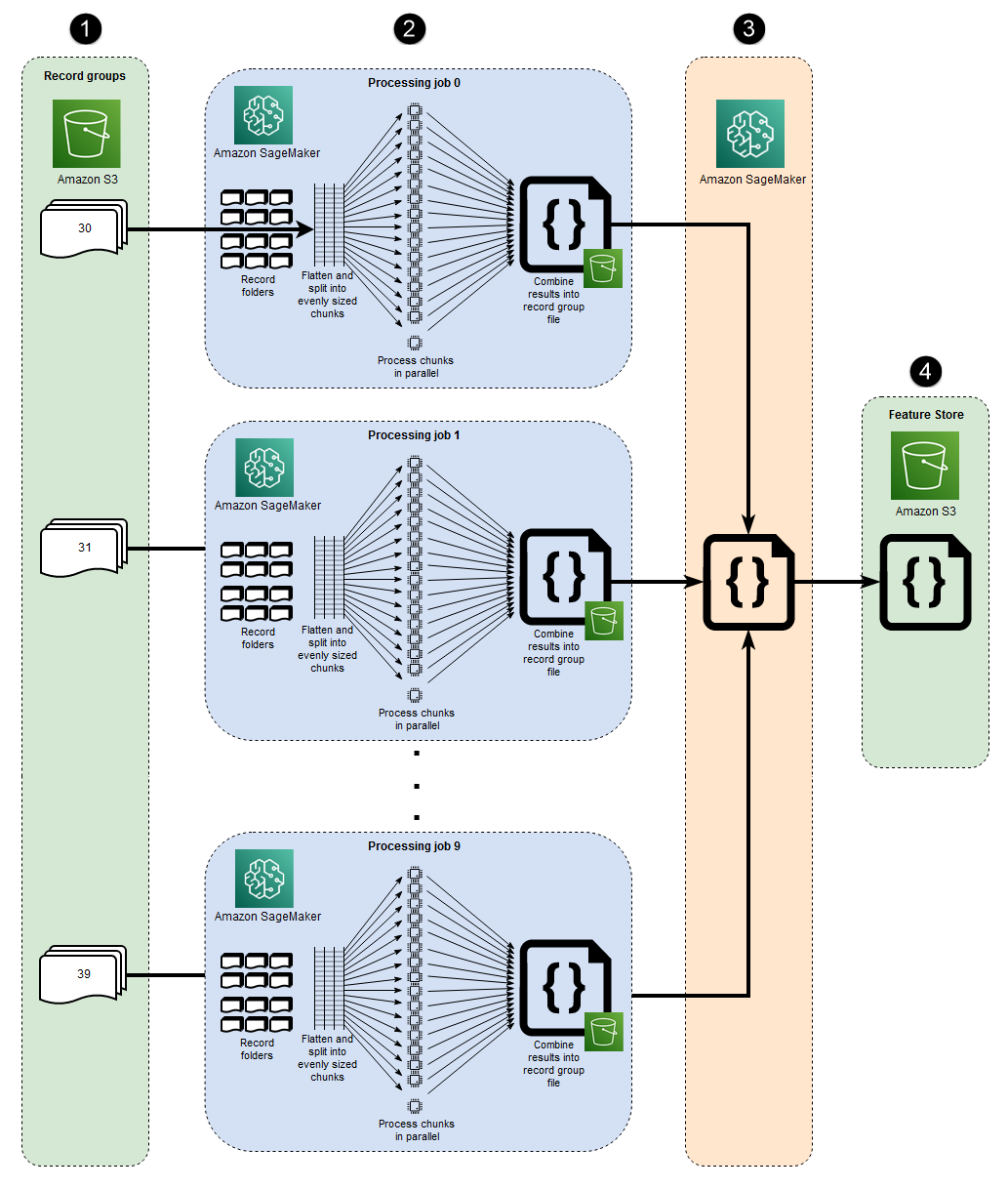
Tuttavia, la suddivisione manuale per gruppo di pazienti significa che è necessario un processo di elaborazione separato per ogni cartella del gruppo di pazienti, come si può vedere nella sezione centrale del diagramma (2), anziché un singolo processo di elaborazione con più istanze. EC2 Poiché i dati di MIMIC-III includono sia file di forme d'onda binarie che file di intestazione basati su testo corrispondenti e che è richiesta la dipendenza dalla libreria wfdbs3_data_distribution_type='FullyReplicated' quando si definisce l'input del processo di elaborazione. In alternativa, se tutti i dati fossero disponibili in un'unica directory e non esistessero dipendenze tra i file, un'opzione più adatta potrebbe essere quella di avviare un singolo processo di Processing con più istanze specificate. EC2 s3_data_distribution_type='ShardedByS3Key' Specificare ShardedByS3Key come indicato dal tipo di distribuzione dei dati di Amazon S3 per gestire automaticamente lo SageMaker sharding dei dati tra le istanze.
L'avvio di un processo di elaborazione per ogni cartella è un modo conveniente per preelaborare i dati, perché l'esecuzione simultanea di più istanze consente di risparmiare tempo. Per ulteriori risparmi in termini di costi e tempi, è possibile utilizzare il microsharding all'interno di ciascun processo di elaborazione.
Microsharding e parallel v CPUs
All'interno di ogni processo di elaborazione, i dati raggruppati vengono ulteriormente suddivisi per massimizzare l'utilizzo di tutte le v disponibili CPUs sull'istanza completamente gestita SageMaker . EC2 I blocchi nella sezione centrale del diagramma (2) illustrano ciò che accade all'interno di ciascun processo di elaborazione principale. Il contenuto delle cartelle cliniche dei pazienti viene appiattito e suddiviso equamente in base al numero di v CPUs disponibili sull'istanza. Dopo la divisione del contenuto della cartella, il set di file di dimensioni uguali viene distribuito su tutti i v CPUs per l'elaborazione. Una volta completata l'elaborazione, i risultati di ogni vCPU vengono combinati in un unico file di dati per ogni processo di elaborazione.
Nel codice allegato, questi concetti sono rappresentati nella sezione seguente del src/feature-engineering-pass1/preprocessing.py file.
def chunks(lst, n): """ Yield successive n-sized chunks from lst. :param lst: list of elements to be divided :param n: number of elements per chunk :type lst: list :type n: int :return: generator comprising evenly sized chunks :rtype: class 'generator' """ for i in range(0, len(lst), n): yield lst[i:i + n] # Generate list of data files on machine data_dir = input_dir d_subs = next(os.walk(os.path.join(data_dir, '.')))[1] file_list = [] for ds in d_subs: file_list.extend(os.listdir(os.path.join(data_dir, ds, '.'))) dat_list = [os.path.join(re.split('_|\.', f)[0].replace('n', ''), f[:-4]) for f in file_list if f[-4:] == '.dat'] # Split list of files into sub-lists cpu_count = multiprocessing.cpu_count() splits = int(len(dat_list) / cpu_count) if splits == 0: splits = 1 dat_chunks = list(chunks(dat_list, splits)) # Parallelize processing of sub-lists across CPUs ws_df_list = Parallel(n_jobs=-1, verbose=0)(delayed(run_process)(dc) for dc in dat_chunks) # Compile and pickle patient group dataframe ws_df_group = pd.concat(ws_df_list) ws_df_group = ws_df_group.reset_index().rename(columns={'index': 'signal'}) ws_df_group.to_json(os.path.join(output_dir, group_data_out))
Una funzione,chunks, viene innanzitutto definita per utilizzare un determinato elenco dividendolo in blocchi di lunghezza di dimensioni uguali n e restituendo questi risultati come generatore. Successivamente, i dati vengono appiattiti tra le cartelle dei pazienti compilando un elenco di tutti i file binari di forme d'onda presenti. Fatto ciò, si ottiene il numero di v CPUs disponibile sull'istanza. EC2 L'elenco dei file binari di forme d'onda viene suddiviso equamente tra questi v CPUs chiamandochunks, e quindi ogni sottolista di forme d'onda viene elaborata sulla propria vCPU utilizzando la classe Parallel di joblib.
Una volta completati tutti i processi di elaborazione iniziali, un processo di elaborazione secondario, mostrato nel riquadro a destra del diagramma (3), combina i file di output prodotti da ciascun processo di elaborazione principale e scrive l'output combinato su Amazon S3 (4).
Strumenti
Strumenti
Python
— Il codice di esempio usato per questo pattern è Python (versione 3). SageMaker Studio: Amazon SageMaker Studio è un ambiente di sviluppo integrato (IDE) basato sul Web per l'apprendimento automatico che ti consente di creare, addestrare, eseguire il debug, distribuire e monitorare i tuoi modelli di machine learning. Puoi eseguire i processi di SageMaker elaborazione utilizzando i notebook Jupyter all'interno di Studio. SageMaker
SageMaker Elaborazione: Amazon SageMaker Processing offre un modo semplificato per eseguire i carichi di lavoro di elaborazione dei dati. In questo modello, il codice di progettazione delle funzionalità viene implementato su larga scala utilizzando i processi di SageMaker elaborazione.
Codice
Il file.zip allegato fornisce il codice completo per questo pattern. La sezione seguente descrive i passaggi per creare l'architettura per questo pattern. Ogni passaggio è illustrato da un codice di esempio contenuto nell'allegato.
Epiche
| Attività | Descrizione | Competenze richieste |
|---|---|---|
| Accedi ad Amazon SageMaker Studio. | Effettua l'onboarding su SageMaker Studio nel tuo account AWS seguendo le istruzioni fornite nella SageMaker documentazione di Amazon. | Scienziato dei dati, ingegnere ML |
| Installa l'utilità wget. | Installa wget se hai effettuato l'onboarding con una nuova configurazione di SageMaker Studio o se non hai mai usato queste utilità in Studio prima. SageMaker Per installarlo, apri una finestra di terminale nella console di SageMaker Studio ed esegui il seguente comando:
| Scienziato dei dati, ingegnere ML |
| Scarica e decomprimi il codice di esempio. | Scarica il
Vai alla cartella in cui hai estratto il file.zip ed estrai il contenuto del
| Scienziato dei dati, ingegnere ML |
| Scarica il set di dati di esempio da physionet.org e caricalo su Amazon S3. | Esegui il notebook | Scienziato dei dati, ingegnere ML |
| Attività | Descrizione | Competenze richieste |
|---|---|---|
| Appiattisci la gerarchia dei file in tutte le sottodirectory. | In set di dati di grandi dimensioni come MIMIC-III, i file sono spesso distribuiti su più sottodirectory anche all'interno di un gruppo principale logico. Lo script deve essere configurato per appiattire tutti i file di gruppo in tutte le sottodirectory, come dimostra il codice seguente.
Nota I frammenti di codice di esempio di questa epopea provengono dal | Scienziato dei dati, ingegnere ML |
| Dividi i file in sottogruppi in base al numero di vCPU. | I file devono essere divisi in sottogruppi o blocchi di dimensioni uguali, a seconda del numero di v CPUs presenti nell'istanza che esegue lo script. Per questo passaggio, puoi implementare un codice simile al seguente.
| Scienziato dei dati, ingegnere ML |
| Parallelizza l'elaborazione dei sottogruppi su v. CPUs | La logica dello script deve essere configurata per elaborare tutti i sottogruppi in parallelo. A tale scopo, utilizzate la
| Scienziato dei dati, ingegnere ML |
| Salva l'output di un singolo gruppo di file su Amazon S3. | Una volta completata l'elaborazione parallela della vCPU, i risultati di ciascuna vCPU devono essere combinati e caricati nel percorso del bucket S3 del gruppo di file. Per questo passaggio, è possibile utilizzare un codice simile al seguente.
| Scienziato dei dati, ingegnere ML |
| Attività | Descrizione | Competenze richieste |
|---|---|---|
| Combina i file di dati prodotti in tutti i processi di elaborazione che hanno eseguito il primo script. | Lo script precedente genera un singolo file per ogni processo di SageMaker elaborazione che elabora un gruppo di file dal set di dati. Successivamente, è necessario combinare questi file di output in un unico oggetto e scrivere un singolo set di dati di output su Amazon S3. Ciò è dimostrato nel
| Scienziato dei dati, ingegnere ML |
| Attività | Descrizione | Competenze richieste |
|---|---|---|
| Esegui il primo processo di elaborazione. | Per eseguire il macrosharding, esegui un processo di elaborazione separato per ogni gruppo di file. Il microsharding viene eseguito all'interno di ogni processo di elaborazione, poiché ogni lavoro esegue il primo script. Il codice seguente mostra come avviare un processo di elaborazione per ogni directory del gruppo di file nel seguente frammento (incluso in).
| Scienziato dei dati, ingegnere ML |
| Esegui il secondo processo di elaborazione. | Per combinare gli output generati dal primo set di processi di elaborazione ed eseguire eventuali calcoli aggiuntivi per la preelaborazione, si esegue il secondo script utilizzando un singolo SageMaker processo di elaborazione. Il codice seguente lo dimostra (incluso in).
| Scienziato dei dati, ingegnere ML |
Risorse correlate
Effettua l'onboarding su Amazon SageMaker Studio utilizzando Quick Start (SageMaker documentazione)
Dati di processo (SageMaker documentazione)
Elaborazione dei dati con scikit-learn (documentazione) SageMaker
Moody, B., Moody, G., Villarroel, M., Clifford, G.D. e Silva, I. (2020). Database delle forme d'onda MIMIC-III
(versione 1.0). PhysioNet. Johnson, A.E.W., Pollard, TJ, Shen, L., Lehman, L.H., Feng, M., Ghassemi, M., Moody, B., Szolovits, P., Celi, L.A. e Mark, R.G. (2016). MIMIC-III
, un database di terapia intensiva accessibile gratuitamente. Dati scientifici, 3, 160035.