기계 번역으로 제공되는 번역입니다. 제공된 번역과 원본 영어의 내용이 상충하는 경우에는 영어 버전이 우선합니다.
테라바이트 규모의 ML 데이터셋의 분산형 피처 엔지니어링에 SageMaker 프로세싱을 사용하기
Chris Boomhower, Amazon Web Services
요약
테라바이트 규모 이상의 많은 데이터 세트는 계층적 폴더 구조로 구성된 경우가 많으며, 데이터 세트에 있는 파일은 때때로 상호 종속성을 공유합니다. 이러한 이유로 기계 학습(ML) 엔지니어와 데이터 과학자는 모델 훈련 및 추론에 사용할 데이터를 준비하기 위해 신중한 설계 결정을 내려야 합니다. 이 패턴은 수동 매크로샤딩 및 마이크로샤딩 기법을 Amazon SageMaker Processing 및 가상 CPU(vCPU) 병렬화와 함께 사용하여 복잡한 빅 데이터 ML 데이터세트에 대한 특성 추출 프로세스를 효율적으로 확장하는 방법을 보여줍니다.
이 패턴은 프로세싱을 위해 매크로샤딩을 여러 시스템에 걸쳐 데이터 디렉터리를 분할하는 것으로 정의하고, 마이크로샤딩은 여러 프로세싱 스레드에 걸쳐 각 시스템의 데이터를 분할하는 것으로 정의합니다. 이 패턴은 PhysioNet MIMIC-III
사전 조건 및 제한 사항
사전 조건
사용자의 데이터세트에 이 패턴을 구현하려는 경우 SageMaker 노트북 인스턴스 또는 SageMaker Studio에 액세스합니다. Amazon SageMaker를 처음 사용하는 경우, AWS 설명서의 Amazon SageMaker 시작하기를 참고하십시오.
PhysioNet MIMIC-III
샘플 데이터로 이 패턴을 구현하려는 경우 SageMaker Studio를 사용합니다. 패턴은 SageMaker Processing을 사용하지만 SageMaker Processing 작업을 실행한 경험이 없어도 됩니다.
제한 사항
이 패턴은 상호 종속적인 파일이 포함된 ML 데이터셋에 매우 적합합니다. 이러한 상호 종속성은 수동 매크로샤딩과 여러 개의 단일 인스턴스 SageMaker Processing 작업을 병렬로 실행할 때 가장 큰 이점을 발휘합니다. 이러한 상호 종속성이 존재하지 않는 데이터셋의 경우 SageMaker Processing의
ShardedByS3Key기능이 매크로샤딩에 대해 더 나은 대안일 수 있습니다. 이 기능은 샤딩된 데이터를 동일한 처리 작업으로 관리되는 여러 인스턴스로 전송하기 때문입니다. 하지만 두 시나리오 모두에서 이 패턴의 마이크로샤딩 전략을 구현하여 인스턴스 vCPU를 가장 잘 활용할 수 있습니다.
제품 버전
Amazon SageMaker Python SDK 버전 2
아키텍처
대상 기술 스택
Amazon Simple Storage Service(S3)
Amazon SageMaker
대상 아키텍처
매크로샤딩 및 분산형 EC2 인스턴스
이 아키텍처에 표시된 10개의 병렬 프로세스는 MIMIC-III 데이터 세트의 구조를 반영합니다. (다이어그램을 단순화하기 위해 프로세스가 타원으로 표시됩니다.) 수동 매크로샤딩을 사용하는 경우 모든 데이터세트에 유사한 아키텍처가 적용됩니다. MIMIC-III의 경우, 최소한의 노력으로 각 환자 그룹 폴더를 개별적으로 처리하여 데이터 세트의 원시 구조를 유리하게 사용할 수 있습니다. 다음 다이어그램에서 레코드 그룹 블록은 왼쪽에 표시됩니다 (1). 데이터가 분산되어 있다는 점을 고려하면 환자 그룹별로 샤딩하는 것이 합리적입니다.
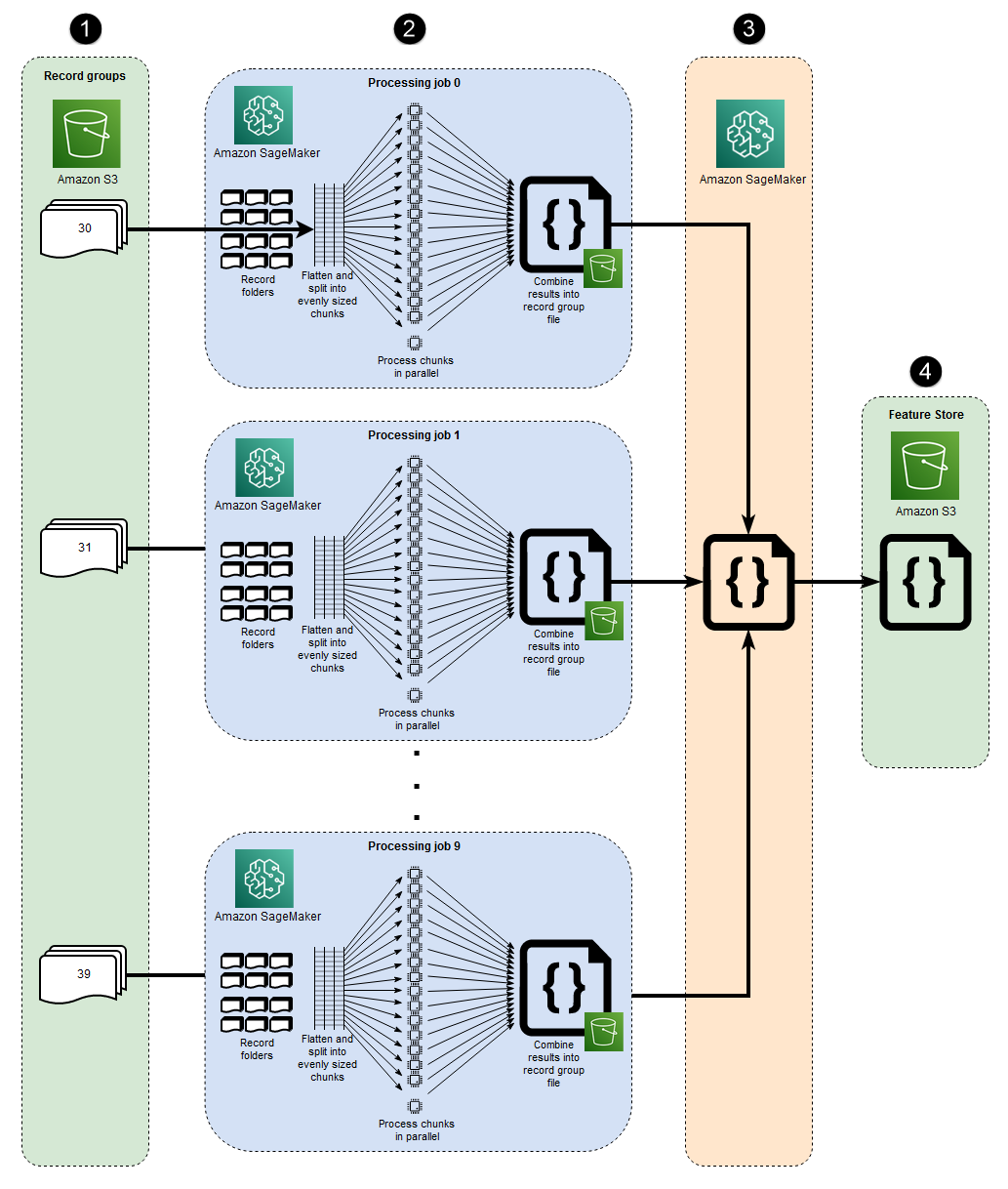
그러나 다이어그램 (2)의 중간 섹션에서 볼 수 있듯이 환자 그룹별로 수동으로 샤딩한다는 것은 여러 EC2 인스턴스가 포함된 단일 처리 작업 대신 각 환자 그룹 폴더마다 별도의 처리 작업이 필요하다는 것을 의미합니다. MIMIC-III의 데이터에는 이진 파형 파일 및 일치하는 텍스트 기반 헤더 파일이 모두 포함되며, 이진 데이터 추출을 위해 wfdb 라이브러리s3_data_distribution_type='FullyReplicated'를 지정하는 것입니다. 또는, 단일 디렉터리에서 모든 데이터를 사용할 수 있고 파일 간에 종속성이 없는 경우에는 여러 EC2 인스턴스 및 s3_data_distribution_type='ShardedByS3Key'를 지정하여 단일 처리 작업을 시작하는 것이 더 적합할 수 있습니다. Amazon S3 데이터 배포 유형으로 ShardedByS3Key 를 지정하면 SageMaker가 인스턴스 전반에서 데이터 샤딩을 자동으로 관리하도록 지시합니다.
여러 인스턴스를 동시에 실행하면 시간이 절약되므로 각 폴더의 처리 작업을 시작하면 데이터를 전처리하는 비용 효율적인 방법이 됩니다. 추가 비용 및 시간을 절약하기 위해 각 처리 작업 내에서 마이크로샤딩을 사용할 수 있습니다.
마이크로샤딩 및 병렬 vCPU
각 처리 작업 내에서 그룹화된 데이터는 SageMaker 완전 관리형 EC2 인스턴스에서 사용 가능한 모든 vCPU의 사용을 극대화하기 위해 추가로 분할됩니다. 다이어그램 (2)의 중간 섹션에 있는 블록은 각 기본 처리 작업에서 발생하는 상황을 나타냅니다. 환자 기록 폴더의 콘텐츠는 인스턴스에서 사용 가능한 vCPU 수에 따라 평탄화되고 균등하게 분할됩니다. 폴더 콘텐츠가 분할되면 동일한 크기의 파일 집합이 처리를 위해 모든 vCPU에 배포됩니다. 처리가 완료되면 각 vCPU의 결과가 각 처리 작업에 대한 단일 데이터 파일로 결합됩니다.
첨부된 코드에서 이러한 개념은 src/feature-engineering-pass1/preprocessing.py 파일의 다음 섹션에 나와 있습니다.
def chunks(lst, n): """ Yield successive n-sized chunks from lst. :param lst: list of elements to be divided :param n: number of elements per chunk :type lst: list :type n: int :return: generator comprising evenly sized chunks :rtype: class 'generator' """ for i in range(0, len(lst), n): yield lst[i:i + n] # Generate list of data files on machine data_dir = input_dir d_subs = next(os.walk(os.path.join(data_dir, '.')))[1] file_list = [] for ds in d_subs: file_list.extend(os.listdir(os.path.join(data_dir, ds, '.'))) dat_list = [os.path.join(re.split('_|\.', f)[0].replace('n', ''), f[:-4]) for f in file_list if f[-4:] == '.dat'] # Split list of files into sub-lists cpu_count = multiprocessing.cpu_count() splits = int(len(dat_list) / cpu_count) if splits == 0: splits = 1 dat_chunks = list(chunks(dat_list, splits)) # Parallelize processing of sub-lists across CPUs ws_df_list = Parallel(n_jobs=-1, verbose=0)(delayed(run_process)(dc) for dc in dat_chunks) # Compile and pickle patient group dataframe ws_df_group = pd.concat(ws_df_list) ws_df_group = ws_df_group.reset_index().rename(columns={'index': 'signal'}) ws_df_group.to_json(os.path.join(output_dir, group_data_out))
chunks 함수는 주어진 목록을 먼저 길이가 n 인 일정한 크기의 덩어리로 나누고 이 결과를 생성기로 반환하여 해당 목록을 소비하도록 정의됩니다. 그런 다음에는 존재하는 모든 이진 파형 파일의 목록을 컴파일하여 환자 폴더 전체에 걸쳐 데이터를 평탄화합니다. 이렇게 하면 EC2 인스턴스에서 사용할 수 있는 vCPU 수가 구해집니다. chunks를 호출하여 이진 파형 파일 목록을 이러한 vCPU에 균등하게 분할한 다음, joblib의 Parallel 클래스
초기 프로세싱 작업이 모두 완료되면 다이어그램 (3)의 오른쪽 블록에 표시된 보조 프로세싱 작업이 각 기본 프로세싱 작업에서 생성된 출력 파일을 결합하고 결합된 출력을 Amazon S3 (4)에 씁니다.
도구
도구
Python
- 이 패턴에 사용되는 샘플 코드는 Python(버전 3)입니다. SageMaker Studio – Amazon SageMaker Studio는 기계 학습 모델을 쉽게 빌드, 훈련, 디버깅, 배포 및 모니터링할 수 있게 해주는 기계 학습을 위한 웹 기반의 통합 개발 환경(IDE)입니다. SageMaker Studio 내에서 Jupyter 노트북을 사용하여 SageMaker 프로세싱 작업을 실행합니다.
SageMaker Processing - Amazon SageMaker Processing은 데이터 처리 워크로드를 실행하는 간소화된 방법을 제공합니다. 이 패턴에서 특성 추출 코드는 SageMaker Processing 작업을 사용하여 대규모로 구현됩니다.
코드
첨부된 .zip 파일은 이 패턴의 전체 코드를 제공합니다. 다음 섹션에서는 이 패턴에 대한 아키텍처를 구축하는 단계를 설명합니다. 각 단계는 첨부 파일의 샘플 코드로 설명됩니다.
에픽
| 작업 | 설명 | 필요한 기술 |
|---|---|---|
| Amazon SageMaker Studio에 액세스합니다. | Amazon SageMaker 설명서에 제공된 지침에 따라 AWS 계정에서 SageMaker Studio에 온보딩합니다. | 데이터 사이언티스트, ML 엔지니어 |
| wget 유틸리티를 설치합니다. | 새로운 SageMaker Studio 구성으로 온보딩했거나 이전에 SageMaker Studio에서 이러한 유틸리티를 사용한 적이 없는 경우 wget을 설치합니다. 이를 설치하려면 SageMaker Studio 콘솔에서 터미널 창을 열고 다음 명령을 실행합니다.
| 데이터 사이언티스트, ML 엔지니어 |
| 샘플 코드를 다운로드하고 압축을 풉니다. | 첨부 파일 섹션에서
.zip 파일을 추출한 위치로 이동하여
| 데이터 사이언티스트, ML 엔지니어 |
| physionet.org에서 샘플 데이터 세트를 다운로드하여 Amazon S3에 업로드합니다. |
| 데이터 사이언티스트, ML 엔지니어 |
| 작업 | 설명 | 필요한 기술 |
|---|---|---|
| 모든 하위 디렉토리의 파일 계층 구조를 평탄화합니다. | MIMIC-III와 같은 대규모 데이터 세트에서는 논리적 상위 그룹 내에서도 파일이 여러 하위 디렉터리에 분산되는 경우가 많습니다. 다음 코드에서 볼 수 있듯이 모든 하위 디렉터리의 모든 그룹 파일을 평탄화하도록 스크립트를 구성해야 합니다.
참고 이 에픽의 예제 코드 조각은 첨부 파일에 제공된 | 데이터 사이언티스트, ML 엔지니어 |
| vCPU 수에 따라 파일을 하위 그룹으로 나눕니다. | 스크립트를 실행하는 인스턴스에 있는 vCPU 수에 따라 파일을 균일한 크기의 하위 그룹 또는 청크로 나누어야 합니다. 이 단계에서는 다음과 비슷한 코드를 구현할 수 있습니다.
| 데이터 사이언티스트, ML 엔지니어 |
| vCPU 전반의 하위 그룹 처리를 병렬화합니다. | 모든 서브그룹을 병렬로 처리하도록 스크립트 로직을 구성해야 합니다. 이렇게 하려면 다음과 같이 Joblib 라이브러리의
| 데이터 사이언티스트, ML 엔지니어 |
| Amazon S3에 단일 파일 그룹 출력을 저장합니다. | 병렬 vCPU 처리가 완료되면 각 vCPU의 결과를 결합하여 파일 그룹의 S3 버킷 경로에 업로드해야 합니다. 이 단계에서는 다음과 비슷한 코드를 사용할 수 있습니다.
| 데이터 사이언티스트, ML 엔지니어 |
| 작업 | 설명 | 필요한 기술 |
|---|---|---|
| 첫 번째 스크립트를 실행한 모든 프로세싱 작업에서 생성된 데이터 파일을 결합합니다. | 이전 스크립트는 데이터 세트의 파일 그룹을 처리하는 각 SageMaker Processing 작업에 대해 단일 파일을 출력합니다. 다음으로, 이러한 출력 파일을 단일 객체로 결합하고 Amazon S3에 단일 출력 데이터 세트를 작성해야 합니다. 이는 첨부 파일에 제공된
| 데이터 사이언티스트, ML 엔지니어 |
| 작업 | 설명 | 필요한 기술 |
|---|---|---|
| 첫 번째 프로세싱 작업을 실행합니다. | 매크로샤딩을 수행하려면 각 파일 그룹에 대해 별도의 프로세싱 작업을 실행합니다. 각 작업에서 첫 번째 스크립트가 실행되므로 각 프로세싱 작업 내에서 마이크로샤딩이 수행됩니다. 다음 코드는 다음 스니펫(
| 데이터 사이언티스트, ML 엔지니어 |
| 두 번째 처리 작업을 실행합니다. | 첫 번째 처리 작업 세트에서 생성된 출력을 결합하고 사전 처리를 위한 추가 계산을 수행하려면 단일 SageMaker Processing 작업을 사용하여 두 번째 스크립트를 실행합니다. 다음 코드는 이를 보여줍니다(
| 데이터 사이언티스트, ML 엔지니어 |
관련 리소스
빠른 시작을 사용한 Amazon SageMaker Studio 온보딩(SageMaker 설명서)
프로세스 데이터(SageMaker 설명서)
scikit-learn을 사용한 데이터 처리(SageMaker 설명서)
Moody, B., Moody, G., Villarroel, M., Clifford, G. D., Silva, I. (2020). MIMIC-III 파형 데이터베이스
(버전 1.0). PhysioNet. Johnson, A. E. W., Pollard, T. J., Shen, L., Lehman, L. H., Feng, M., Ghassemi, M., Moody, B., Szolovits, P., Celi, L. A., Mark, R. G. (2016). MIMIC-III(무료로 이용할 수 있는 중환자 치료 데이터베이스)
Scientific Data, 3, 160035.
첨부
이 문서와 관련된 추가 콘텐츠에 액세스하려면 attachment.zip 파일의 압축을 풉니다.