本文為英文版的機器翻譯版本,如內容有任何歧義或不一致之處,概以英文版為準。
使用 SageMaker Processing 對 TB 級 ML 資料集進行分散式特徵工程
Chris Boomhower,Amazon Web Services
Summary
許多 TB 級或更大的資料集通常由階層式資料夾結構組成,而且資料集中的檔案有時會共用相互依存性。因此,機器學習 (ML) 工程師和資料科學家必須做出深思熟慮的設計決策,以準備此類資料以進行模型訓練和推論。此模式示範如何結合 Amazon SageMaker Processing 和虛擬 CPU (vCPU) 平行處理使用手動巨集分片和微分技術,以有效率地擴展複雜大數據 ML 資料集的功能工程程序。
此模式將巨集碎片定義為跨多部機器處理的資料目錄分割,並將微碎片定義為跨多個處理執行緒分割每部機器上的資料。模式透過使用 Amazon SageMaker 搭配 PhysioNet MIMIC-III
先決條件和限制
先決條件
如果您想要為自己的資料集實作此模式,請存取 SageMaker 筆記本執行個體或 SageMaker Studio。如果您是第一次使用 Amazon SageMaker,請參閱 AWS 文件中的開始使用 Amazon SageMaker。
SageMaker Studio,如果您想要使用 PhysioNet MIMIC-III
範例資料實作此模式。 模式使用 SageMaker Processing,但不需要任何執行 SageMaker Processing 任務的經驗。
限制
此模式非常適合包含相互依存檔案的 ML 資料集。這些相互依存性受益於手動巨集分割和平行執行多個單一執行個體 SageMaker Processing 任務。對於不存在此類相互依存性的資料集,SageMaker Processing 中的
ShardedByS3Key功能可能是巨集碎片的更佳替代方案,因為它會將碎片資料傳送至由相同處理任務管理的多個執行個體。不過,您可以在這兩種情況下實作此模式的微分策略,以充分利用執行個體 vCPUs。
產品版本
Amazon SageMaker Python SDK 第 2 版
架構
目標技術堆疊
Amazon Simple Storage Service (Amazon S3)
Amazon SageMaker
目標架構
巨集控制和分散式 EC2 執行個體
此架構中表示的 10 個平行程序會反映 MIMIC-III 資料集的結構。(程序會以省略符號表示,以簡化圖表。) 當您使用手動巨集分片時,類似的架構會套用至任何資料集。在 MIMIC-III 的情況下,您可以盡可能分別處理每個病患群組資料夾,藉此善用資料集的原始結構。在下圖中,記錄群組區塊會出現在左側 (1)。鑑於資料的分散式本質,依病患群組碎片是合理的。
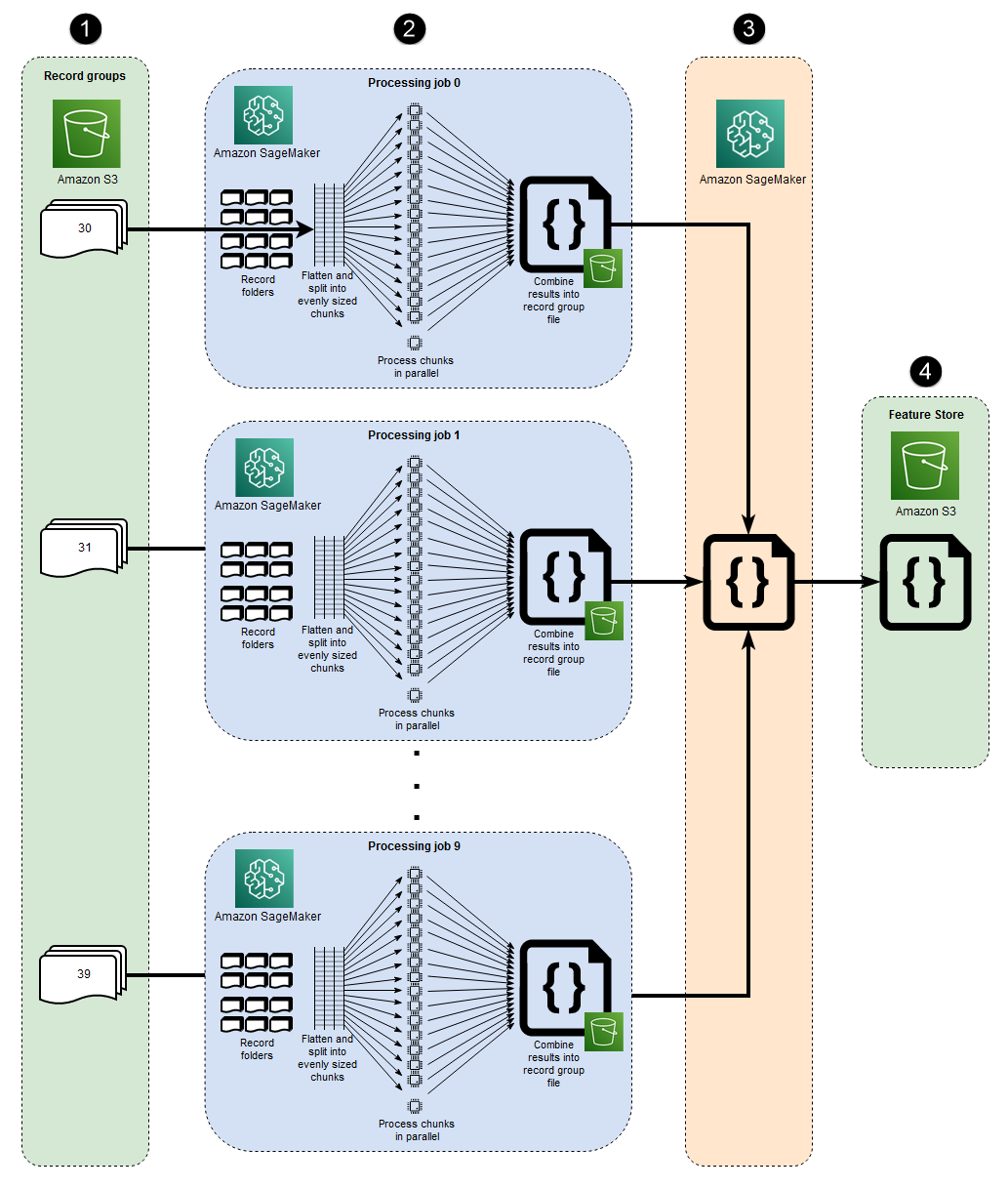
不過,依病患群組手動分片表示每個病患群組資料夾都需要單獨的處理任務,如圖表 (2) 中間區段所示,而不是具有多個 EC2 執行個體的單一處理任務。由於 MIMIC-III 的資料同時包含二進位波形檔案和相符的文字型標頭檔案,而且需要依賴 wfdb 程式庫s3_data_distribution_type='FullyReplicated'何時定義處理任務輸入。或者,如果所有資料在單一目錄中可用,而且檔案之間不存在相依性,則更合適的選項可能是啟動具有多個 EC2 執行個體且s3_data_distribution_type='ShardedByS3Key'指定的單一處理任務。將 指定ShardedByS3Key 為 Amazon S3 資料分佈類型,會指示 SageMaker 自動管理跨執行個體的資料分片。
為每個資料夾啟動處理任務是一種經濟實惠的方式來預先處理資料,因為同時執行多個執行個體可節省時間。為了節省額外的成本和時間,您可以在每個處理任務中使用微分。
微分和平行 vCPUs
在每個處理任務中,分組的資料會進一步分割,以最大限度地使用 SageMaker 全受管 EC2 執行個體上所有可用的 vCPUs。圖表 (2) 中間區段中的區塊描述了每個主要處理任務中發生的情況。會根據執行個體上可用的 vCPUs 數量,將病患記錄資料夾的內容扁平化並平均分割。分割資料夾內容後,平均大小的檔案集會分散到所有 vCPUs以進行處理。處理完成時,每個 vCPU 的結果會合併為每個處理任務的單一資料檔案。
在連接的程式碼中,這些概念會呈現在 src/feature-engineering-pass1/preprocessing.py 檔案的下一節中。
def chunks(lst, n): """ Yield successive n-sized chunks from lst. :param lst: list of elements to be divided :param n: number of elements per chunk :type lst: list :type n: int :return: generator comprising evenly sized chunks :rtype: class 'generator' """ for i in range(0, len(lst), n): yield lst[i:i + n] # Generate list of data files on machine data_dir = input_dir d_subs = next(os.walk(os.path.join(data_dir, '.')))[1] file_list = [] for ds in d_subs: file_list.extend(os.listdir(os.path.join(data_dir, ds, '.'))) dat_list = [os.path.join(re.split('_|\.', f)[0].replace('n', ''), f[:-4]) for f in file_list if f[-4:] == '.dat'] # Split list of files into sub-lists cpu_count = multiprocessing.cpu_count() splits = int(len(dat_list) / cpu_count) if splits == 0: splits = 1 dat_chunks = list(chunks(dat_list, splits)) # Parallelize processing of sub-lists across CPUs ws_df_list = Parallel(n_jobs=-1, verbose=0)(delayed(run_process)(dc) for dc in dat_chunks) # Compile and pickle patient group dataframe ws_df_group = pd.concat(ws_df_list) ws_df_group = ws_df_group.reset_index().rename(columns={'index': 'signal'}) ws_df_group.to_json(os.path.join(output_dir, group_data_out))
函數 chunks會先定義為透過將指定清單分割為平均大小的長度區塊n ,並將這些結果傳回為產生器,來使用指定的清單。接下來,透過編譯存在的所有二進位波形檔案清單,將資料扁平化到病患資料夾。完成後,會取得 EC2 執行個體上可用的 vCPUs 數量。透過呼叫 將二進位波形檔案的清單平均分割到這些 vCPUschunks,然後使用 joblib 的平行類別
當所有初始處理任務完成時,次要處理任務會顯示在圖表 (3) 右側的 區塊中,結合每個主要處理任務產生的輸出檔案,並將合併的輸出寫入 Amazon S3 (4)。
工具
工具
Python
– 用於此模式的範例程式碼為 Python (第 3 版)。 SageMaker Studio – Amazon SageMaker Studio 是適用於機器學習的 Web 型整合式開發環境 (IDE),可讓您建置、訓練、偵錯、部署和監控機器學習模型。您可以在 SageMaker Studio 中使用 Jupyter 筆記本來執行 SageMaker Processing 任務。
SageMaker Processing – Amazon SageMaker Processing 提供執行資料處理工作負載的簡化方法。在此模式中,使用 SageMaker Processing 任務大規模實作特徵工程程式碼。
Code
連接的 .zip 檔案提供此模式的完整程式碼。下一節說明為此模式建置架構的步驟。每個步驟都由附件中的範例程式碼說明。
史詩
| 任務 | 描述 | 所需的技能 |
|---|---|---|
| 存取 Amazon SageMaker Studio。 | 遵循Amazon SageMaker 文件中提供Amazon SageMaker Studio。 | 資料科學家、ML 工程師 |
| 安裝 wget 公用程式。 | 如果您使用新的 SageMaker Studio 組態加入,或從未在 SageMaker Studio 中使用這些公用程式,請安裝 wget。 若要安裝,請在 SageMaker Studio 主控台中開啟終端機視窗,然後執行下列命令:
| 資料科學家、ML 工程師 |
| 下載並解壓縮範例程式碼。 | 在附件區段中下載
導覽至您解壓縮 .zip 檔案的資料夾,並解壓縮
| 資料科學家、ML 工程師 |
| 從 physionet.org 下載範例資料集,並將其上傳至 Amazon S3。 | 在包含 | 資料科學家、ML 工程師 |
| 任務 | 描述 | 所需的技能 |
|---|---|---|
| 扁平化所有子目錄的檔案階層。 | 在 MIMIC-III 等大型資料集中,檔案通常分佈於多個子目錄,即使在邏輯父群組內也是如此。您的指令碼應設定為在所有子目錄中扁平化所有群組檔案,如下列程式碼所示。
注意 此範例程式碼片段的範例來自 檔案,該 | 資料科學家、ML 工程師 |
| 根據 vCPU 計數將檔案分成子群組。 | 根據執行指令碼的執行個體上存在vCPUs 數量,檔案應分為大小均勻的子組或區塊。在此步驟中,您可以實作類似如下的程式碼。
| 資料科學家、ML 工程師 |
| 平行處理跨 vCPUs的子群組。 | 指令碼邏輯應設定為平行處理所有子群組。若要這樣做,請使用 Joblib 程式庫的
| 資料科學家、ML 工程師 |
| 將單一檔案群組輸出儲存至 Amazon S3。 | 當平行 vCPU 處理完成時,每個 vCPU 的結果應合併並上傳至檔案群組的 S3 儲存貯體路徑。在此步驟中,您可以使用類似如下的程式碼。
| 資料科學家、ML 工程師 |
| 任務 | 描述 | 所需的技能 |
|---|---|---|
| 合併執行第一個指令碼的所有處理任務所產生的資料檔案。 | 先前的指令碼會為每個 SageMaker Processing 任務輸出單一檔案,該任務會從資料集處理一組檔案。 接著,您需要將這些輸出檔案合併為單一物件,並將單一輸出資料集寫入 Amazon S3。這是在 檔案中示範的,該
| 資料科學家、ML 工程師 |
| 任務 | 描述 | 所需的技能 |
|---|---|---|
| 執行第一個處理任務。 | 若要執行巨集分割,請為每個檔案群組執行個別的處理任務。Microsharding 會在每個處理任務內執行,因為每個任務都會執行您的第一個指令碼。下列程式碼示範如何在下列程式碼片段 (包含在 中
| 資料科學家、ML 工程師 |
| 執行第二個處理任務。 | 若要合併第一組處理任務產生的輸出並執行任何額外的預先處理運算,您可以使用單一 SageMaker Processing 任務來執行第二個指令碼。下列程式碼示範這一點 (包含在 中
| 資料科學家、ML 工程師 |
相關資源
使用 Quick Start 加入 Amazon SageMaker Studio (SageMaker 文件)
處理資料 (SageMaker 文件)
使用 scikit-learn 的資料處理 (SageMaker 文件)
Moody、B.、Moody、G.、Villanerroel、M.、Clifford、G. D. 和 Silva、I. (2020)。MIMIC-III 波形資料庫
(1.0 版)。PhysioNet。 Johnson, A. E. W., Pollard, T. J., Shen, L., Lehman, L. H., Feng, M., Ghassemi, M., Moody, B., Sezovits, P., Celi, L. A., & Mark, R. G. (2016)。MIMIC-III,可免費存取的關鍵護理資料庫
。科學資料,3, 160035。
附件
若要存取與本文件相關聯的其他內容,請解壓縮下列檔案: attachment.zip